科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-07-02
2021年7月2日,英国约翰英纳斯研究所(John Innes Centre)冯小琦课题组在国际顶级学术期刊 Science 杂志发表了题为“Nurse cell- derived small RNAs define paternal epigenetic inheritance in Arabidopsis”的研究长文,揭示了植物生殖细胞特异DNA甲基化重编程的分子机制。
研究背景
被子植物生殖发育过程雄性生殖细胞经历DNA甲基化重编程:除了整体DNA甲基化水平发生变化外,基因组的一些局部位点会发生de novo DNA 甲基化(de novo DNA methylation)。DNA甲基化重编程过程对植物生殖发育至关重要。冯小琦实验室2018年的研究结果表明,雄性生殖细胞DNA甲基化除了发生在转座子(TE)外,也会出现在基因(gene)上,从而调控靶标基因表达,影响减数分裂过程。虽然生殖细胞中de novo DNA甲基化是受RNA介导的DNA甲基化途径(RNA-directed DNA methylation pathway, 简称RdDM)调控,但是,RdDM途径在植物各个组织中广泛表达且主要用于沉默转座子。因此,RdDM如何作用在生殖细胞基因上仍然是一个谜。研究这个机制不仅影响我们对植物的跨代表观遗传的理解至关重要,而且直接影响我们对RdDM途径是如何在细胞水平作用的认识。
主要研究结果
为了解雄性生殖细胞中特异DNA甲基化形成的原因,研究人员首先分离了雄性减数分裂细胞(male meiocyte,图1),并构建了小RNA (sRNA)文库。sRNA-seq的分析结果发现,减数分裂细胞中绝大多数(~94%)24-nt sRNA只来源于大约800个位点,这与体细胞大为不同,表明减数分裂细胞sRNA发生了重编程。进一步分析发现这800个位点绝大多数与转座子重合,且被高度DNA甲基化,因此研究人员把它们命名为HyperTE。然而,令人不解的是,对于发生在基因区间上的DNA 甲基化位点(命名为MetGene),却几乎检测不到24-nt sRNA的存在。那么,MetGene 上的DNA甲基化又是如何形成的呢?
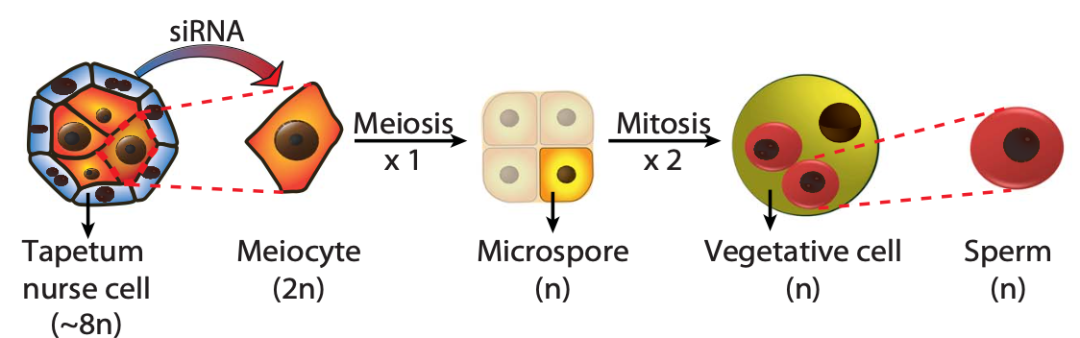
图1:拟南芥雄性细胞减数分裂示意图
深入分析发现,HyperTE 与MetGene 位点存在一定的序列相似性。当24-nt sRNA 允许3个以下碱基错配时,MetGene上就有显著的sRNA富集信号。接下来,CRISPR技术敲除HyperTE的实验证实了来源于HyperTE的24-nt sRNA通过允许错配的方式反式(trans) 催化MetGene 上的DNA甲基化。
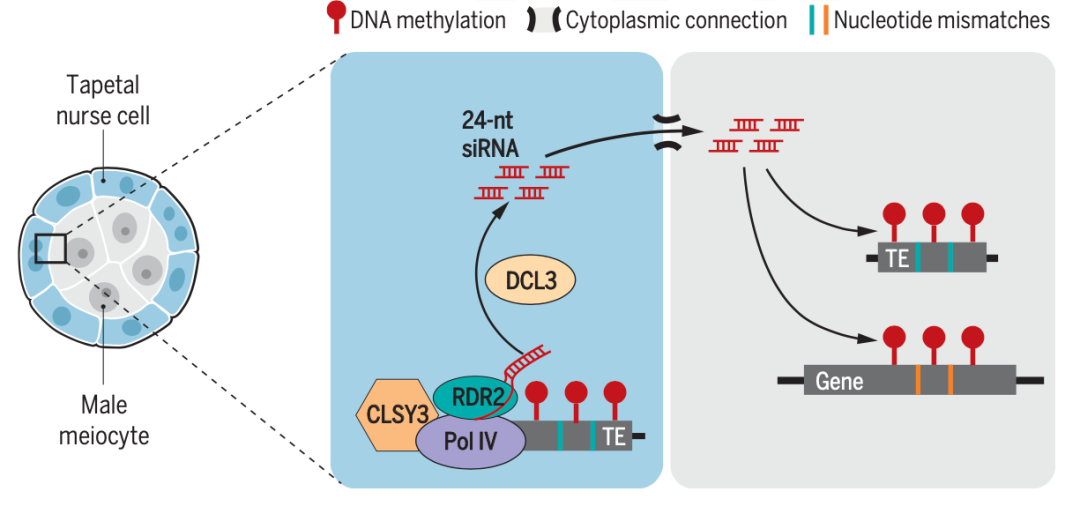
RdDM需要Pol IV 和 Pol V两个重要的RNA聚合酶参与。Pol IV 被招募到富含DNA甲基化的区域催化24-nt sRNA前体的合成,24-nt sRNA 被装载在AGO 蛋白后,被招募到Pol V转录产生的长非编码RNA处,最终促成DNA甲基化。因此,RdDM 是一个正反馈调节过程。减数分裂细胞中MetGene位点存在较高DNA甲基化,却鲜有24-nt sRNA产生,暗示着减数分裂细胞中Pol IV 可能不工作。那么如果减数分裂细胞自己不产生24-nt sRNA ,其来源于何处呢?研究人员把目光转向了环绕在减数分裂细胞周边的绒毡层细胞上(tapetum),因为先前研究表明,绒毡层细胞与减数分裂细胞存在大量胞间连丝,这为sRNA的转移提供了理论上可能。
为了验证这一假设,研究人员首先构建了绒毡层细胞特异表达启动子驱动的GFP的株系(pTP::GFP,图2),利用流式细胞分选技术(FACS),分离出了纯和的绒毡层细胞。绒毡层细胞的DNA甲基组测序结果表明, 与减数分裂细胞类似,绒毡层细胞在HyperTE位点的DNA甲基化程度很高。sRNA-seq 数据进一步显示绒毡层细胞24nt-sRNA的分布模式和减数分裂细胞十分相似。以上结果为支持sRNA 从绒毡层细胞转移到减数分裂细胞的假设提供了初步证据。为了进一步证明绒毡层细胞产生的24-nt RNA 可以转移到减数分裂细胞催化DNA 甲基化,作者构建了pTP::RDR2 rdr2遗传镶嵌株系(RDR2 负责将单链Pol IV 转录本转换成双链,是24-nt sRNA 合成不可或缺的因子。此株系中,只有绒毡层细胞具有产生24-nt sRNA 的能力)。pTP::RDR2 rdr2 减数分裂细胞甲基组测序结果显示,相较于rdr2突变体中DNA 甲基化完全丢失,该株系在HyperTE 和MetGene位点的DNA甲基化能恢复到野生型水平。以上结果证明了绒毡层细胞产生的24-nt sRNA能够充分介导减数分裂细胞的DNA 甲基化。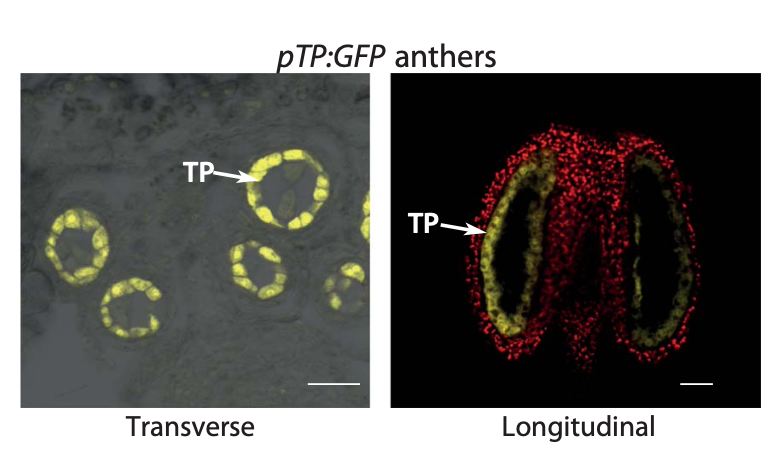
图2:绒毡层细胞特异表达启动子驱动的GFP的株系表达图谱,TP: 绒毡层细胞
那么导致绒毡层细胞出现大量24nt-sRNA的原因是什么?研究人员发现,HyperTE位点与已经发表的染色质重组蛋白CLSY3 依赖的位点高度重合,而与CLSY1,CLSY2 和CLSY4 依赖的位点很少关联。CLSY1/2/3/4是CLSY 家族四个蛋白,他们都能够与Pol IV 直接互作, 决定Pol IV被招募到基因组的特定位置。基于此,研究人员猜想减数分裂细胞中24-nt sRNA 是由CLSY3介导产生的。为了证实这一猜想,研究人员首先分析了不同细胞和组织CLASSY家族转录本的表达情况,发现只有CLSY3在绒毡层细胞中高度表达。pCLSY3::Venus-CLSY3报告株系结果显示,CLSY3蛋白在雄蕊中的绒毡层细胞中特异表达,这与转录本分析结果一致。进一步分析结果显示,野生型减数分裂细胞24-nt sRNA富集在CLSY3依赖的位点,而不在CLSY1,CLSY2 或CLSY4 依赖的位点。当CSLY3缺失(而非其他CLSY家族成员缺失)才会导致减数分裂细胞中HyperTE 和MetGene 位点上24-nt sRNA 丰度和DNA甲基化的丢失。综合以上结果,证明CSLY3是导致生殖细胞特异DNA甲基化出现的根本原因,也再次印证了减数分裂细胞中24-nt sRNA来源于绒毡层细胞。
clsy3突变体精子的甲基组数据显示,HyperTE 和MetGene 上DNA甲基化缺失; 而pTP::RDR2 rdr2精子中上述位点DNA甲基化程度与野生型相当。以上结果表明绒毡层细胞产生的24-nt sRNA 不仅驱动减数分裂细胞中DNA甲基化,同样也介导在精子中的DNA 甲基化(该阶段绒毡层细胞已经消亡)。文章的最后,研究还发现,绒毡层细胞产生的24-nt sRNA负责维持逆转录转座子Gypsy1 的沉默状态。
一图解文
本研究系统地揭示了拟南芥雄性生殖发育过程中,绒毡层细胞表达的CLSY3在HyperTE 位点催化产生大量24-nt sRNA,随后被转移到减数分裂细胞中,介导HyperTE和MetGene上DNA甲基化的形成,最终调控靶标基因表达和维持生殖细胞基因组的稳定性。这一发现不仅诠释了RdDM通过特别招募Pol IV 及sRNA碱基错配形成细胞特异的甲基化模式的分子机制,而且翻开了被子植物跨代表观遗传机制的新篇章。
主要作者介绍
英国约翰英纳斯研究所冯小琦研究员为本论文的通讯作者(http://www.jic.ac.uk/people/xiaoqi-feng/),博士后龙金成,James Walker 和佘文静为文章共同第一作者。本研究由欧洲科学院(European Research Council, ERC),英国生物技术与生物科学研究委员会 (Biotechnology and Biological Sciences Research Council, BBSRC)以及约翰英纳斯基金会 (JIC Foundation)资助。
来源:PlantBiotech 植物生物学
原文链接:http://mp.weixin.qq.com/s?__biz=MzI5NTk2MTcyOA==&mid=2247498178&idx=2&sn=d9a57b96f6530141dbddf9e71e21bd8b
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

DNA到底能不能预测外貌?

自然中不存在的蛋白质也能人造了!撼动材料医药界
Nat Plants:植物如何防晒?抑制DNA甲基化
营养与健康所阐述血液中细胞特异性的DNA甲基化改变

复旦大学揭示减数分裂细胞中组蛋白去甲基化酶底物特异性及调控染色质浓缩的分子机制
科学家阐述血液中细胞特异性的DNA甲基化改变
非对称聚合物材料可制造灵活多变的软机器人
CCS Chemistry | 纳米卫星胶囊探针,同时检测细胞内端粒酶和DNA甲基化水平

营养与健康所阐述血液中细胞特异性的DNA甲基化改变
演化:“最奇怪动物”身份揭秘