科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-10-01
前言在进行微生物多样性分析时,大家一定会做α,β多样性分析。
通俗来讲,α多样性就是样本内的物种多样性。
β多样性是指在地区尺度上,物种组成沿着某个梯度方向从一个群落到另一个群落的变化率。
即沿着某一环境梯度,物种替代的速率、物种周转率等。
排序的过程是将样品或微生物物种排列在一定的空间, 使得排序轴能够反映一定的生态梯度 这些排序方法又可以分成间接梯度排序(indirect gradient analysis)和直接梯度排序(direct gradient analysis)。
间接梯度排序又叫非约束性排序;寻求潜在的或在间接的环境梯度来解释物种数据的变化包括PCA,PCoA,NDMS,直接排序又叫约束性排序;它是指在特定的梯度上(环境轴) 上探讨物种的变化情况;方法包括 RDA, CCA, db-RDA。
排序分析(Ordination analysis)。
排序(ordination)的过程就是在一个可视化的低维空间或平面重新排列这些样本,使得样本之间的距离最大程度地反映出平面散点图内样本之间的关系信息。
db-RDA 介绍distance-based redundancy analysis (db-RDA) 是目前在微生物领域应用的最为广泛的环境因子分析,该分析方法内置在R中的vegan包中。
相信大家一定都知道vegan包,该R包是进行生态学(包括微生物多样性分析)研究的必备神器!
vegan包中提供了所以基本排序分析的方法,可以说是一包在手搞定所有!
关于vegan包的详细介绍,请大家查看vegan包的官方文档。
本文还会给大家介绍另一款vegan的开挂版本,ggvegan的介绍与使用,ggvegan相当于在vegan软件包中内置了ggplot2, 绘制的图片比用vegan直接绘图更好看!
微生物环境因子分析要进行微生物环境因子分析,我们需要两个文件,一个是微生物多样性的OTU 表格,另一个就是你所有样品的环境因子数据。
比如,你进行土壤微生物研究,这时候你就需要知道你所测土壤的C,N,P,K等化学元素含量以及不同样地的气候信息等等,总之,在分析之前可以多准备些环境因子数据,后期我们还可以对这些环境因子进行共线性,以及环境因子与数据拟合优良性判断。
数据均一化首先看看我们准备的OTU表格以及环境因子数据结构(图1):OTU表格(图2):环境因子数据读取完数据之后,我们要把OTU的横轴和纵轴调换位置,然后把OTU表格也要进行hellinger转化,使数据均一性更好。
并把环境因子进行log转化,以减少同一种环境因子之间本身数值大小造成的影响。
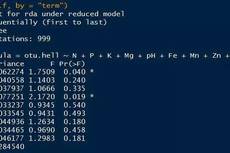
RDA和CCA模型筛选数据都进行均一化之后,我们要进行RDA和CCA的模型筛选。
先用species-sample资料做DCA分析看分析结果中Lengths of gradient的第一轴中的Axis lengths大小,如果大于4.0,就应该选CCA,如果在3.0-4.0之间,选RDA和CCA均可,如果小于3.0,RDA的结果要好于CCA。
本例中,我们数据的Axis lengths 大小为0.63859,所有我们应该选择RDA进行分析!
(图3)差膨胀因子分析在筛选完RDA和CCA分析后,我们需要利用方差膨胀因子分析,对所有环境因子进行共线性分析。
我们要依次删掉最大的变量,也就是删除掉共线性的环境因子,直到所有的变量都小于10。
检测最低AIC值最后我们要用step模型检测最低AIC值,在这一步中该模型会自动筛选出最优的环境因子。
当“none”位于最顶端时意味着改模型筛选结束,位于none值上方的环境因子即为与OTU拟合最好的环境因子。
来源:宏基因组
原文链接:http://mp.weixin.qq.com/s?__biz=MzUzMjA4Njc1MA==&mid=2247501706&idx=3&sn=e1550a1882891fc03af15301ebaf94f9
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
微生物学: 一个微生物培养基配方数据库

夯实我会党建工作 党总支书记讲党课
微生物环境因子分析(RDA/db-RDA)-ggvegan包
微生物学: 生命早期的抗生素使用可能会影响发育
国家微生物科学数据中心

环保部与联合国儿童基金会合作项目在我会启动

黑河中下游防风固沙功能时空变化及影响因子分析
口腔微生物会破坏阴道微生物平衡

版纳植物园海拔梯度根际微生物研究获进展
微生物所发表中国微生物组数据平台