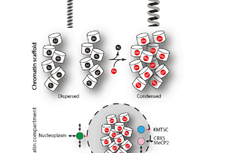
人们很早就知道染色质结构与基因表达的调控密切相关(如常染色质/异染色质)。随着近几年染色质构象捕捉技术的发展,人们了解到细胞核内的DNA存在着多重结构调节机制,例如A/B染色质区室(compartment)和拓扑结构域(TAD)。它们的大小分别在百万碱基和十万碱基的左右。然而由于测序深度和技术条件的限制,Hi-C很难达到更高的分辨率,这也限制了人们分析关于单个基因,以及启动子(P)、增强子(E)等调控元件之间的相互作用。
近日,Molecular Cell杂志发表背靠背文章Resolving the 3D Landscape of Transcription-Linked Mammalian Chromatin Folding和Ultrastructural Details of Mammalian Chromosome Architecture。它们发展了一种新的染色质构象捕捉技术,名为Micro-C。Micro-C可以达到单个核小体长度(~200bp)左右的分辨率,因此可以观察到许多更为精细的染色质结构。
Micro-C和Hi-C在实验步骤上最大的不同,在于Micro-C先对核小体上缠绕的DNA做交联,而后用MNase切断核小体之间的连接DNA。以Tjian和Darzacq实验室的文章为例,他们对38个小鼠干细胞进行Micro-C测序,总共获得约26亿个reads。在相似的测序深度下,20kb精度的Micro-C图谱与Hi-C结果很相似,但是在100至20kb的精度之间,Micro-C展示了Hi-C所不具备的精细结构,例如很多有关基因启动子和增强子的结构域、条带和点圈。从Hi-C可以得到约6000个DNA环(loop),即高度相互作用的一对区域。而Micro-C可以得到约30000个。
在证实了Micro-C具备可重复性和高精度之后,作者展开了进一步的分析。相比Hi-C,Micro-C独特的地方主要在于它可以探查到调控元件之间的相互作用,如EP,PP等。其中有些相互作用形成了比传统TAD小的结构域。研究者通过隔离分值的分析得到了大约13万个这样的结构域边界。这些边界通常有着活跃转录的基因和动态的核小体结构,而这与之前所了解的,以CTCF和Cohesin为标志的TAD边界有很大不同。
作者利用回归模型分析了这些结构域边界和表冠遗传特征的关联性。他们发现,CTCF和Cohesin的确在决定边界位置上是最重要的,然而决定这些边界强度的却主要是转录活跃的标志物,如H3K4me3修饰和染色质开放度等等。他们进一步发现,这些边界彼此间也存在很大差别,于是将其分为了五类:转录相关;干细胞特异性增强子;一般增强子;抑制型;CTCF-Cohesin相关型。它们各有不同的化学修饰。
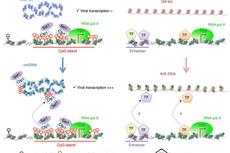
作者选取了一些代表性的区域,他们发现,不仅启动子、增强子和CTCF之间可以发生相互作用,在聚梳抑制区还可以观察到显著的“巢状”环。CTCF的相互作用范围较长,而Pol II介导的相互作用则较短。基因附近的相互作用越紧密,其转录活性也常常越高。为了进一步证明转录对染色质折叠的影响,作者利用药物抑制转录。他们对比观察到,TAD等大尺度的结构没有发生变化,然而基因附近的相互作用条纹大大减弱了,证明了这些条纹团确实是由转录过程导致的。
总的来说,这两篇文章显示了哺乳动物的染色质相互作用还有更多的精细结构等待开发。更高精度的数据能帮助人们理解这些结构的形成和对基因转录的影响。
原文链接:https://doi.org/10.1016/j.molcel.2020.03.002https://doi.org/10.1016/j.molcel.2020.03.003
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652482974&idx=3&sn=acc0381ecc6ab6779e335510b87de062&chksm=84e23e2ab395b73cf7444813f879f5e5f7cf4d5c8792d19ede02dc6f539222b72e5093c1ab5d#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn