科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-07-17
来源:宏基因组
写在前面本系列按原文4幅组图,共分为4节。本文是第一节,模式图与主坐标轴分析。
先回顾一下图1的内容。
图1. 水稻根系微生物随时间变化吗?实验设计与群落整体结构(主坐标轴PCoA)分析
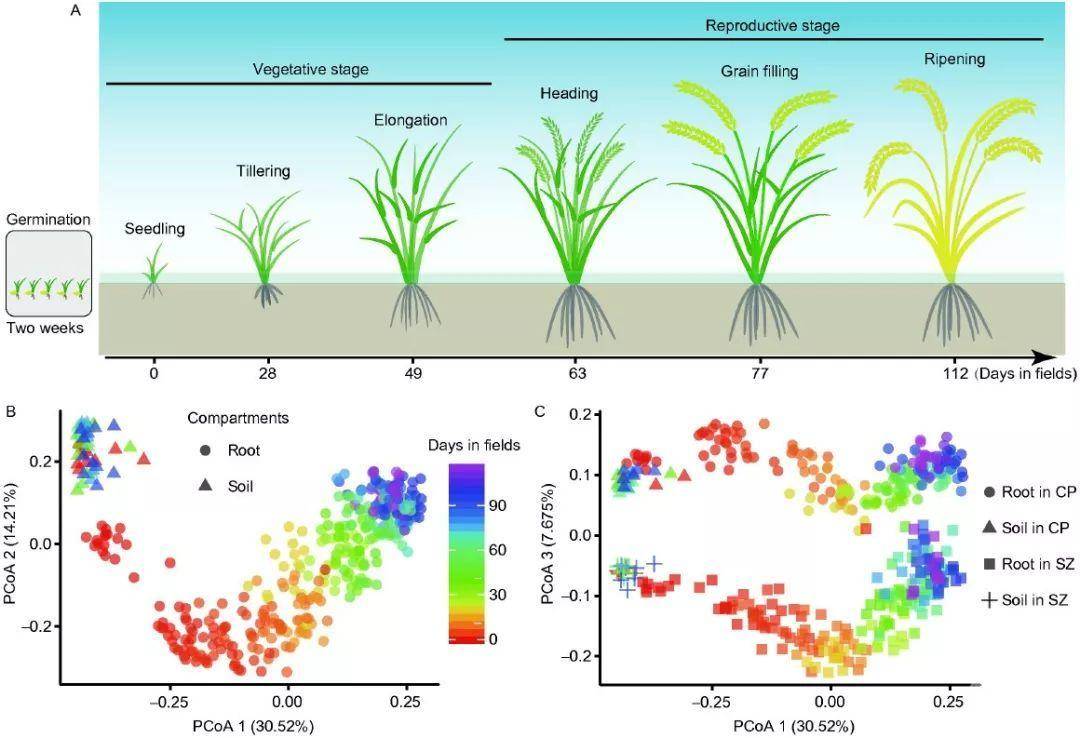
图1. 田间水稻微生物组随生育时间变化
A. 水稻全生育期根系微生物组实验设计模式图(以水稻日本晴和IR24为材料,并分别种植于昌平和上庄两地,CP代表北京昌平农场,SZ代表北京海淀上庄)
B-C. 主坐标轴分析(PCoA)展示水稻微生物组随时间变化,其中微生物群落结构主要在第1/2轴上随时间变化(B),而不同土壤类型主要在第3轴上明显分开(C)
方法说明:图1A采用Aodbe Illustrator手绘,具体方法可参考youbute上使用AI绘制花的教程,https://www.youtube.com/watch?v=WSxemBP-gZQ。
图1B/C是基于Bray-Curtis距离进行的PCoA分析,采用散点图展示,并按时间顺序填充彩虹色,按不同compartment设置形状。图1B展示PCo1/2轴,组间最大差异为不同compartment与时间梯度上的变化。图1C展示PCo1/3轴,可进一步看到1轴的差异与时间变化一致,而3轴可以很好分开不同地点。
绘制模式图,工具有非常多的选择。有的大神用PPT可以画出美到极致的模式图,有的人用喜欢用PS。在这里我们推荐用AI,因为它是学术界矢量图绘制排版神器,没有之一。
AI是Adobe Illustrator软件的缩写,对于各种软件生成的矢量图,混合编辑,轻松满足各类杂志的所有要求。没有基础的小伙伴可以学习下文:
教师节献礼 - 文章用图的修改和排版
本文中是用AI绘制了水稻不同生育期的矢量图,绘制还是要求有一定基础和思路,以及AI的基本使用技巧才能完成。
个人感觉软件操作比较容易学习,难点是如何设计合理的模式图,及基本的绘图技术。设计、构思、修改都是极花时间的,只有原作者对自己的项目理解最深刻,最容易构思较好的模式图,具体的绘制建议拿草图找会画画的同学合作。
图1B/C. 主坐标轴分析主坐标轴分析需要两个输入文件,一个是实验设计,即样本与分组(时间)对应表。另一个是样本间的距离矩阵(距离矩阵可通过OTUs直接计算获得,详见《扩增子分析流程》中第6节)。
下面开始数据筛选与绘图,请按文末说明下载数据和代码,在Rstudio中即可重现。
打开代码文件,设置工作目录
# Fig.1 PCoA每次绘图前(list=ls())清空工作环境,防止有之前的旧变量导致绘图结果中有错误
# clean enviroment object加载本分析需要的包,主要包括生态统计的vegan包和一年引用超7000次的绘图神器ggplot2,没有安装过此包的朋友请使用Rstudio中Packages页面手动安装
# load related packages设置图形输出的基本样式,即theme,每个人都有自己的风格,这是我的习惯风格,如字体7号是Nature杂志推荐的字号。经验积累一个自己的theme,可以为后期AI排版节约很多时间,也有自己的风格。
# Set ggplot2 drawing parameter, such as axis line and text size, lengend and title size, and so on.读取实验设计并筛选
现在的实验一般都有特别多的组,如本研究包括两品种、两地点,再乘以时间点近100组,500个样品。肯定会有人为造成的异常样本,如何通过实验分析结果、配合实验记录排除人为错误或不可控因素造成的影响呢?那就是仔细观察数据和实验记录,综合判断后筛选样本,是十分必要的。
我们的分析中也发现了两个时间点样本的异常,根据农场的记录对应,发现了这两个点分别进行了大规模的施肥和除虫。将确实对分析影响较大的异常样本和组剔除后再次分析。
# Public file 1. "design.txt" Design of experiment读取距离矩阵
# PCoA bray_curtis距离矩阵与实验设计的交叉筛选
这步是非常必要的,因为实验设计在不断的分析中,会删除一些异常样本。而OTU表中也会有些样本数据量过低、过高而被删除。大量样本时不完全对应,需要进行交叉筛选保持实验设计与数据矩阵一致。
# subset matrix and design主坐轴分析cmdscale
结果有很多信息,主要提取结果中的坐标位置和各轴的解析差异的比例
# cmdscale {stats}, Classical multidimensional scaling (MDS) of a data matrix. Also known as principal coordinates analysis添加样品组信息:合并PCoA坐标与实验设计
points = cbind(points, sub_design[match(rownames(points), rownames(sub_design)), ])绘图:散点图展示样品坐标,XY轴标签显示可解析差异的比例,按时间序列为分组连序着色,按不同取样部分显示不同形状
# plot PCo 1 and 2预览结果并保存:注意图片输出PDF格式,可以在AI中进一步编辑文件和线条,图片大小4 x 2.5适合大多数杂志的1/2栏大小
p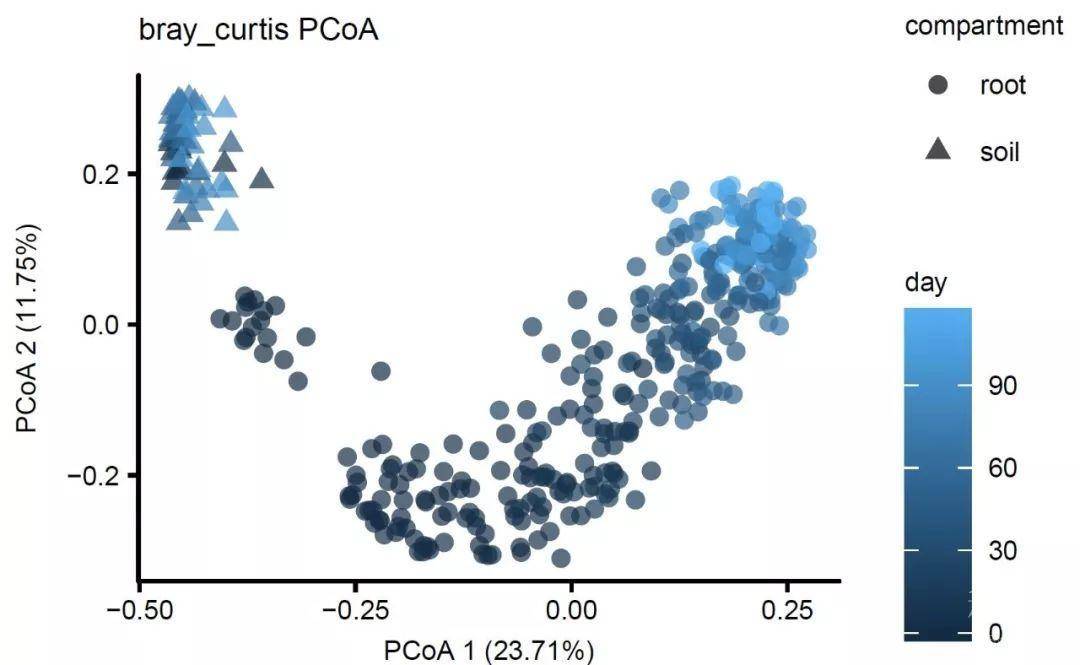
这个图是把时间变化展示出来了,但默认的深蓝到浅蓝,太低调,不够妖艳。我个人喜欢R ggplot2绘图另一个原因是喜欢它的彩虹色系统。
彩虹色绘制第1/2主轴的时间变化规律
# scale_color_gradientn 按多种颜色连续着色,如彩虹色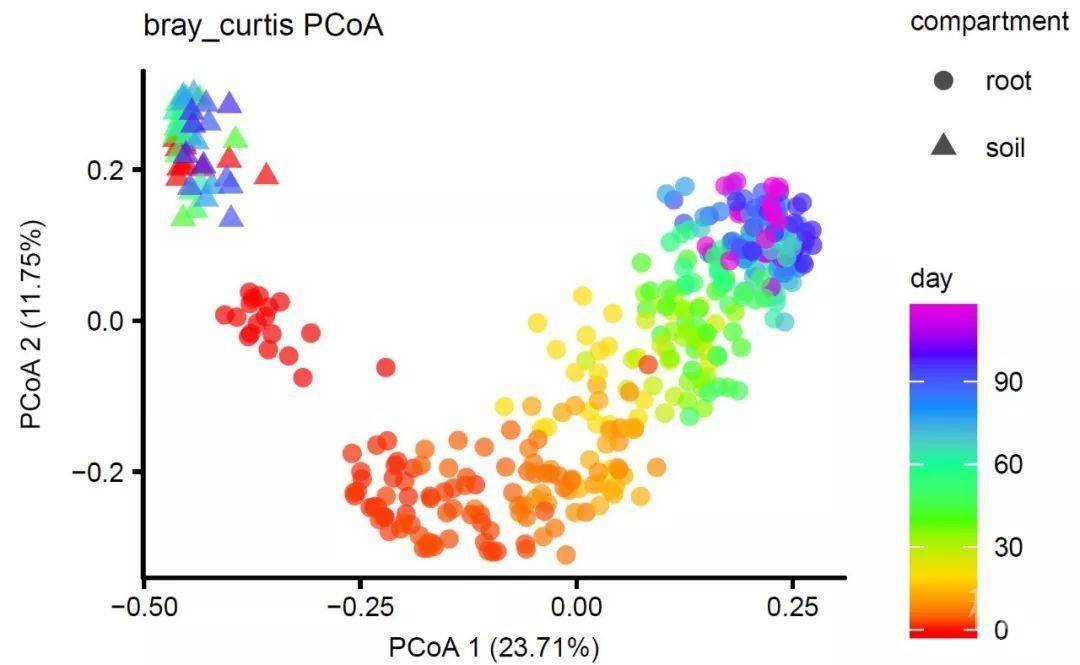
现在看时间变化是不是清楚多了,纯个人感觉,也行有的朋友不喜欢这种风格,更有一些杂志社严格要求不允许使用彩虹色。那就要看你根据杂志要求和自己的表现目标调整了,上面代码注释行中也提了众多可调整的函数方案可选。
PCoA有众多维度,通常看1/2轴可解析的差异也最大。但有时你研究的目标末必是最大差异,可以进一步往下找,如1/3,1/4,3/4轴。
这里我们绘制1/3轴
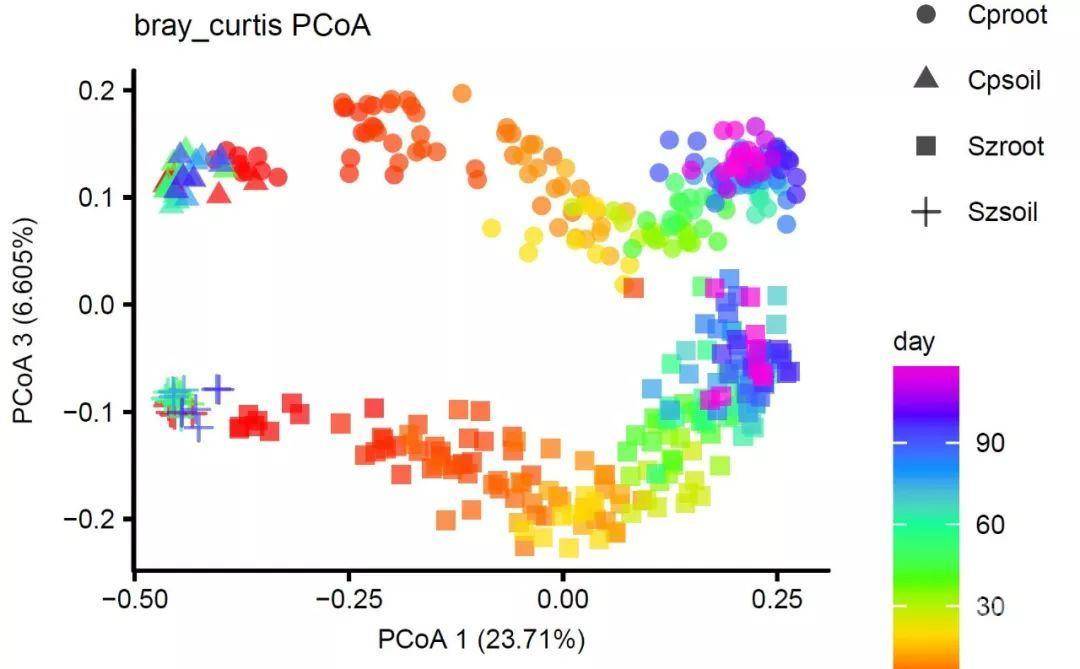
采用1/3轴组合,即展示出时间序列变化,又展示出了两地点昌平(Cp)和上庄(Sz)间的明显差异。
来源:meta-genome 宏基因组
原文链接:https://mp.weixin.qq.com/s?__biz=MzUzMjA4Njc1MA==&mid=2247491159&idx=3&sn=da13d92f07c39f6a37b86dd7801a507e&chksm=fab9f4e6cdce7df07617fd967c53c6e9cceffe81d7c90573559a653d6073c392437b8ac099ae#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
人类直立行走的时间再度提前
时间序列预测法
水文时间序列分析

8月,聚焦浦东科协 盛飨学术盛宴

3D打印技术使移动工厂成现实:大大缩短病人等待时间

人体各部位微生物组时间序列分析Moving Pictures

学术交流 | 资源型城市长时间序列土壤含水量变化分析——以锡林浩特市为例

人体各部位微生物组时间序列分析Moving Pictures
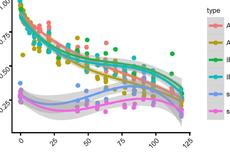
水稻微生物组时间序列分析2b-散点图拟合

“时间贫穷”:中国人的时间都去哪了?