科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-07-21
来源:iNature
iNature

来源:Plant_ihuman iNature
原文链接:https://mp.weixin.qq.com/s?__biz=MzU3MTE3MjUyOA==&mid=2247512209&idx=5&sn=431c15e402f3bec8444117780cebf255&chksm=fce6c34ecb914a588218403561616c1a0b6db25b579ae34e4b32e3054321a8cd406a633feafd#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

北京农林科学院玉米基因组编辑团队在碱基编辑技术方面再获突破

高彩霞团队建立新型可预测多核苷酸删除基因组编辑系统

中科院遗传发育所高彩霞研究组建立新型、可预测的多核苷酸删除基因组编辑系统

遗传发育所建立新型可预测多核苷酸删除基因组编辑系统

【Cell】重磅!拟南芥为模型研究人类神经系统疾病的发病机制!
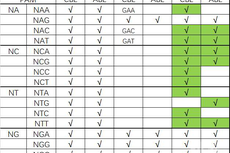
北京市农林科学院玉米基因组编辑团队大幅拓展基因组编辑范围

高彩霞研究组建立新型可预测多核苷酸删除基因组编辑系统
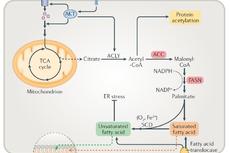
Nat Rev MCB | 代谢调控与细胞增殖
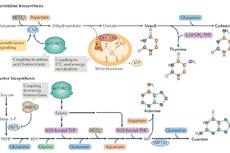
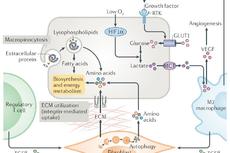

实验表明“原生汤”增稠将加速早期生命进化

量子计算机首次识别出单个核苷酸