科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-04-26
来源:生命科学
作者:王木桂 , 朱健康
摘要:多基因编辑技术对于基因家族和通路的研究以及作物改良具有十分重要的意义。利用CRISPR-Cas系统在植物中进行多基因编辑需要同时表达Cas基因和多个sgRNA,组成一个多转录元件系统。为了简化多基因编辑体系,人们利用RNA自剪切工具和植物内源的RNA加工机制把多转录元件改造成双转录元件和单转录元件。该文介绍了植物多基因编辑体系由繁至简不断优化过程中的经典案例,同时对存在的问题及发展的方向进行讨论。
CRISPR (clustered regularly interspaced short palindromic repeats)/Cas (CRISPR-associated gene)来源于细菌的免疫系统,主要由CRISPR序列元件与Cas基因组成。在细菌中,CRISPR元件是一类独特的DNA重复序列簇,由一些高度保守的重复序列(repeats)和间隔序列(spacer)相间排列组成,而Cas基因是位于CRISPR序列附近一些高度保守的家族基因,具有核酸酶活性,可对DNA序列进行剪切[1]。该免疫系统在工作时,CRISPR序列转录形成CRISPR RNA (crRNA),与另一种转录形成的反式作用型crRNA (trans-activating crRNA, tracrRNA)部分区域配对形成二元复合体,然后该二元复合体一起引导具有非特异核酸酶活性的CAS蛋白切割与crRNA匹配的DNA序列,通常为侵入细菌体内的外来DNA序列,如噬菌体、质粒等等[2]。为方便在真核生物中应用,工程化的CRISPR系统将crRNA与tracrRNA融合在一起形成单一的sgRNA (single guide RNA)[3-4]。在植物中进行基因编辑通常需要同时构建sgRNA表达盒和Cas基因表达盒,其中sgRNA表达盒比较小,一般由长约300 bp的U3或U6等pol Ⅲ (RNA polymerase Ⅲ)启动子驱动,以连续6个以上的T碱基(poly T, pT)终止即可,而Cas基因的表达则通常由pol Ⅱ (RNA polymerase Ⅱ)类型的35S或Ubi等强启动子驱动,以PolyA或NOS等终止子结束[5-6]。
在动物或人类细胞里进行多基因编辑时可以共转染多个含有sgRNA表达盒的质粒,甚至直接混合注射其RNA,而在植物体内进行基因编辑往往需要利用农杆菌介导等方法实现稳定的遗传转化,当进行单次多基因定点编辑时必须把Cas基因表达盒、筛选标记表达盒以及多个sgRNA表达盒共同装配到一个载体上,组成一个多转录元件系统(multi- component transcriptional unit system, MCTU)(图 1)。
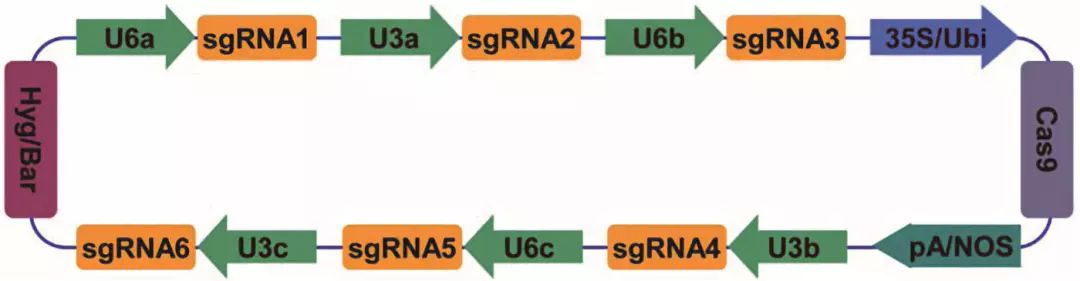
pA/NOS: poly A或NOS等终止子;Hyg/Bar:潮霉素或草铵膦等抗性基因。
图 1 MCTU系统
1 MCTU——经典的植物多基因编辑系统
2015年,刘耀光研究组开发了一套经典的适用于单子叶和双子叶植物的多基因编辑载体系统,可利用一个载体同时表达多个sgRNA单元[7]。为避免过多重复序列可能导致的片段重组丢失、基因沉默等风险,该研究在水稻和拟南芥中分别选用4个pol Ⅲ类型的启动子:OsU3、OsU6a、OsU6b、OsU6c (对水稻),或AtU3b、AtU3d、AtU6-1、AtU6-29 (对拟南芥),组成若干个不同的sgRNA表达盒同时靶向不同的目标位点。为方便组装这一套复杂的载体系统,研究人员巧妙地设计了一系列的PCR扩增引物和酶切黏性末端,可通过Gibson片段装配法或Golden Gate连接法将所有sgRNA模块一次性克隆到包含有Cas9表达盒和筛选标记表达盒的双元载体上。另外,载体上的克隆位点携带了ccdB致死基因,克隆失败的菌株在LB板上长不出来,从而进一步提高了载体构建效率。在这项研究中,研究人员构建了20个靶向水稻单基因和多基因的敲除载体,其中靶向目标最多的一个质粒搭载了8个sgRNA表达盒。另外,还构建了4个靶向拟南芥1~3个基因的载体。实验结果表明,水稻所有被编辑的46个位点平均突变率高达85.4%,并且大多数突变类型为双等位突变或纯合突变,而在拟南芥的基因敲除效率也达到35.6%,充分说明了这套多基因编辑系统的高效性。
2016年,朱健康研究组亦报道了类似构架的MCTU系统用于拟南芥的多基因编辑[8]。该研究从拟南芥中克隆了3个pol Ⅲ启动子:AtU6-26、AtU3b和At7SL-2,分别使用两次构成一个6敲系统靶向拟南芥PYR/PYL基因家族的6个基因位点,在T1代植株检测发现各靶点的突变率达13%~93%,并且能获得6基因同时突变的株系。
MCTU系统作为第一代植物多基因编辑体系已经广泛用于植物多基因位点的敲除,由于每个sgRNA均由单独的启动子驱动表达,相互之间不干扰,系统内各sgRNA单元的基因敲除效率一般与相应的单基因敲除载体无异,但也正是因为这样导致系统构架复杂,通常由一系列中间载体组成,构建起来程序繁琐且费时费力,往往需要有经验的技术人员才能顺利完成。
2 TCTU——一个表达盒搭载多个sgRNA单元
为了简化复杂的MCTU系统,科研人员尝试把多个sgRNA表达盒整合成一个,这样加上Cas9基因表达盒可以把多基因编辑体系简化为双转录元件系统(two component transcriptional unit system, TCTU)(图 2)。当利用一个启动子转录一串sgRNA阵列,各sgRNA单元需要经过特定的加工剪切机制进行分离释放,于是一系列具有RNA加工剪切功能的元件(RNA processing elements, RPE)进入了人们的视线。tRNA (transfer RNA)是生物体内广泛存在的用于携带并转运氨基酸的一类小分子核糖核酸,由一条长70~90个核苷酸并折叠成三叶草形的短链组成。在真核细胞核内,tRNA的前体由RNase P和RNase Z识别并于特定位点切割,被切除的5′和3′冗余序列没有序列限制性[9-10]。根据这些特征,2015年,杨益农研究组利用植物内源的tRNA加工成熟机制开发了多顺反子tRNA-gRNA系统(polycistronic tRNA-gRNA, PTG),可以利用一个pol Ⅲ启动子同时转录生成多个sgRNA,在这一过程中不需要添加外源的RNA加工剪切工具[11]。该研究选择在植物体内广泛存在的甘氨酸转运tRNA (tRNAGly)元件(相应的DNA序列长度为77 bp)作为间隔,将数量不等的sgRNA单元(最多达8个)组装成一系列的PTG系统,通过原生质体转化靶向水稻多个位点,结果表明各PTG系统的DNA切割效率为4%~45%,同时,水稻稳定转化实验的结果也表明各靶向位点的突变率最高可达100%。
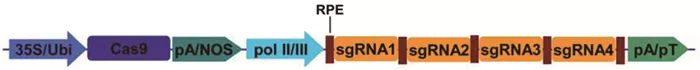
RPE: RNA加工剪切元件;pol Ⅱ/Ⅲ: RNA聚合酶Ⅱ或Ⅲ相关的启动子;pA/NOS: poly A或NOS等终止子;pT: poly T终止序列。
图 2 TCTU系统
与tRNA类似的常用于组装多基因编辑器的RNA加工剪切工具还有Csy4、核酶(ribozyme, RZ)等。Csy4是来源于细菌的RNA剪切酶,在细菌的免疫系统里,当CRISPR基因簇被转录成前体(pre-crRNA),Csy4就通过序列与结构的特异性把该前体分割成单个成熟的crRNA[12]。Csy4 RNA酶及其附属的仅20 bp长度的特异识别切割序列被用于构建人类细胞的多基因编辑器并证实其有效性[13]。核酶是一类具有催化活性的RNA小分子,可通过催化靶位点RNA链中磷酸二酯键的断裂,特异性地剪切底物RNA分子。两种名为Hammerhead和HDV (hepatitis delta virus)的短链核酶序列(其相应DNA序列的长度分别为43 bp和68 bp)曾被用来组装RGR (ribozyme-gRNA-ribozyme)系统,分别设置在sgRNA序列的5′端和3′端,用于从pol Ⅱ启动子转录出来的sgRNA前体中精确地切割分离成熟的sgRNA序列,最先于2014年在酵母及人类细胞的基因编辑实验中得到验证[14-15]。另一种具有15 bp识别位点的Hammerhead核酶元件也被成功地用于从pol Ⅱ转录物中切割释放1~2个sgRNA单元,水稻转化实验表明其生成的sgRNA引导Cas9靶向编辑目标位点的效率最高达100%[16]。
2017年,Voytas研究组在番茄原生质体细胞中系统地比较了pol Ⅲ构架的MCTU体系和pol Ⅱ构架的TCTU体系在多基因编辑上的效率[17]。在MCTU体系中,两个pol Ⅲ启动子AtU6和At7SL,被组装成三套双基因敲除系统。而在TCTU体系中,3种不同类型的RNA加工剪切工具tRNA、Csy4和核酶元件,各组成一个双基因敲除系统。由于常用的35S启动子已被用来驱动Cas9基因的表达,为避免在一个T-DNA区上重复使用相同启动子造成基因沉默的风险,在TCTU体系中选择了来自丁香属黄叶卷曲病毒(Cestrum Yellow Leaf Curling Virus)的pol Ⅱ类型启动子CmYLCV驱动sgRNA阵列的转录,因为该启动子适用于很多类型的作物,并且其驱动基因表达的能力在双子叶植物里不亚于35S,而在单子叶里不亚于ZmUbi [18]。研究人员对转化后的原生质体进行高通量测序表明,使用Csy4和tRNA组装的TCTU系统所产生的突变率大概是MCTU系统的两倍,3个MCTU系统之间没有显著差异,而利用核酶元件组成的TCTU系统进行基因编辑的效率最低。随后,上述3个TCTU系统被扩大到同时编辑8个靶位点,结果表明,Csy4构架的效率最高,tRNA次之,而核酶系统的表现最差[17]。核酶元件有很多种,由于在上述研究中所使用的核酶的识别位点仅为15 bp,可能会对细胞体内相同或类似的转录序列也进行识别切割从而产生毒性,最终影响基因编辑的效率。尽管不同的研究对核酶元件的评价不一,但Voytas的上述研究充分证明了TCTU体系在多基因编辑中的效率可以优于经典的MCTU体系,而且结构简单,构建更加方便。另外,在TCTU系统里由于RNA加工剪切工具的使用,sgRNA的表达不必受限于pol Ⅲ启动子,众多各具特色的pol Ⅱ启动子为其提供了更加灵活的选择。
3 Cpf1——天然的TCTU系统
Cpf1 (CRISPR from Prevotella and Francisella 1,又名为Cas12a)是一种新型的class 2-type V CRISPR系统,与属于class 2-type II的Cas9相比具有诸多独特的特征:它识别富含A/T碱基的PAM序列(TTN或TTTN),DNA切割位点位于PAM的远端并产生黏性末端。另外,在CRISPR/Cpf1系统中,成熟的crRNA仅由一条很短的直接重复序列(direct repeat, DR, 20~21 bp)和引导序列(guide, 22~23 bp)组成,不需要反式作用型tracrRNA的辅助;更重要的是,Cpf1蛋白兼具DNA剪切酶和RNA剪切酶的功能,可以把转录出来的crRNA前体加工剪切成成熟型crRNA[19-20]。这样,Cpf1蛋白自身就具备了类似TCTU系统中RNA加工剪切元件的功能。利用Cpf1的这种特性可以把多个crRNA单元直接串联在一起表达,转录后Cpf1蛋白自身可以把单个sgRNA切割分离并分别靶向不同的位点,这一过程不需要像Cas9多基因编辑系统那样添加额外的RNA处理元件。这一构思率先通过动物细胞实验和小鼠体内实验取得成功。在体外和体内实验中,研究人员利用AsCpf1 (Cpf1 from Acidaminococcus)分别构建了由4个和3个DR-guide单元组成的crRNA阵列进行多基因编辑,发现各靶点的突变效率与单基因编辑系统相当,阵列内不同guide的排列次序不存在位置效应;另外,在研究中还发现由成熟型DR和guide序列组成的阵列比序列更长的非成熟型阵列具有更高的效率[21]。
2017年,朱健康研究组利用FnCpf1 (Cpf1 from Francisella novicida)和LbCpf1 (Cpf1 from Lachnospiraceae bacterium ND2006)对水稻进行单基因和多基因敲除测试,发现上述两类Cpf1只需一条非常短的DR序列(20~21bp)加上22~24 bp的guide序列即可在植物体内实现单基因敲除;更重要的是,把多个成熟型DR-guide单元直接串联,只需要一个pol Ⅲ启动子驱动即可简单高效地实现多基因敲除。实验中测试了由4个DR-guide单元组成的crRNA短阵列分别对水稻RLK和CYP81A家族的四个基因进行编辑,结果表明,各靶点的敲除效率达到40%~ 75%[22]。Cpf1的开发利用拓展了CRISPR系统在动植物中的应用,利用Cpf1可以组装成一个天然的TCTU系统,为多基因编辑提供了一个新利器(图 3)。但是相对于Cas9,目前Cpf1在动植物中尚未得到广泛的使用,在一些双子叶植物(如拟南芥)中基因编辑效果不佳,仍然需要进一步优化改良。
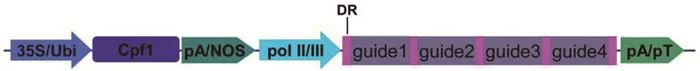
R:直接重复序列;pol Ⅱ/Ⅲ: RNA聚合酶Ⅱ或Ⅲ相关的启动子;pA/NOS: poly A或NOS等终止子;pT: poly T终止序列。
图 3 利用Cpf1构建的TCTU系统
4 STU——只有一个表达盒的多基因编辑器
TCTU系统借助外源或内源的RNA加工工具已经实现了把多个sgRNA单元组装到一个表达盒里,但仍然独立于Cas9或Cpf1等核酸酶的表达盒之外,能否进一步把这两个基本的表达盒也整合到一起,这一设想首先在人类细胞的基因编辑中实现。2015年,Naito研究组构建了由上文提到的Hammerhead和HDV核酶元件组装而成的包含sgRNA的RGR模块,并把RGR模块内嵌于Cas9表达盒的5′UTR (untranslated region)区,由一个pol Ⅱ启动子同时驱动sgRNA和Cas9,转录后sgRNA可由核酶元件分离,而Cas9的mRNA则进入下一步的翻译阶段,这样就构成了一种由单个转录元件组成的基因编辑系统(single transcriptional unit system, STU)[23]。该研究利用包含1~2个RGR模块的STU系统对HEK 293细胞系进行了转化实验,结果表明STU系统能有效地切割靶位点;但是在上述研究中所构建STU系统的缺陷在于,由于RGR模块被设置在Cas9的5′端,有可能由于ATG碱基的存在而导致mRNA被提前翻译,所以,在这个系统中还有必要在Cas9的5′端添加一段内部核糖体进入位点序列(internal ribosome entry site, IRES)以保证Cas9准确地起始翻译。2016年,张勇研究组设计的STU系统在植物的基因编辑中也取得了成功[24]。研究人员先利用eYFP报告基因在水稻原生质体中测试了多种linker序列对STU系统内基因转录和翻译的影响,发现当设置有核酶元件时,以一段人工合成的polyA序列(pA)作linker能保证eYFP报告基因的正常表达而不受核酶剪切的影响。随后,研究人员构建了以下构架的STU系统:pol Ⅱ-Cas9-pA-RZsite-sgRNA1- RZsite-sgRNA2-RZ,在水稻中进行单基因和双基因位点的敲除实验,结果表明,靶位点的突变率达到53.8%~100%。在这个构架的设计上,由于sgRNA模块被设置在Cas9完整表达盒的3′端,所以不需要添加额外的IRES序列去保证Cas9的起始翻译。
谢卡斌研究组于2018年开发了分别基于Cas9和Cpf1的STU系统[25],研究人员发现在UBI10启动子的5′UTR区域包含一段非常保守的内含子序列,该序列的边界上具有在mRNA成熟过程中内含子被切除时识别的基序5′-GU-A-AG-3′,理论上可以把其中的内含子序列换成sgRNA模块,这样在用UBI10启动子驱动Cas9时,位于5′UTR区域GU-A-AG基序内的sgRNA被识别为内含子而切割分离,而作为外显子的Cas9则正常表达。在装载Cas9的STU系统里,当需要串联多个sgRNA时,各单元之间可用tRNA元件连接,而在装载Cpf1的STU系统里,由于Cpf1蛋白自身兼具RNA酶活性,各guide单元之间可直接用其DR序列作间隔。水稻转化实验表明,这两套STU系统均能有效地进行单基因和多基因位点敲除。
以上论述的三套STU系统在设计上各有特色:Naito研究组把sgRNA模块设置在Cas9基因的5′UTR区域,并且利用Hammerhead和HDV核酶元件进行分离,但需要添加额外的IRES元件保证Cas9的起始翻译;张勇研究组充分利用了polyA终止子不完全终止转录的特征把sgRNA模块设置在Cas9表达盒的3′端,同样利用核酶元件进行分离;而谢卡斌研究组则利用了一段保守的内含子剪切基序用于内嵌sgRNA序列,利用tRNA元件实现各个sgRNA模块的分离。这些STU系统尽管各有各的特点,共同的特征是仅需要一个pol Ⅱ启动子就能同时实现Cas9 (或者Cpf1)和sgRNA的转录,在结构上比TCTU系统更简单,但其串联的单个和多个sgRNA模块之间还必需借助额外添加的各种RNA加工剪切元件才能有效地分离释放(图 4)。

RPE: RNA加工剪切元件;pA/NOS: poly A或NOS等终止子。
图 4 STU系统
5 SSTU——把多基因编辑体系简化到极致
TRBO (Tobacco mosaic virus RNA-based overex-pression)是一个来源于烟草花叶病毒的过表达系统,常用于植物体内大量表达目的基因。在一项研究中,Cody等[26]本计划利用TRBO系统在烟草中大量表达sgRNA,却发现在植物体内sgRNA的5′和3′端均能容忍一定的多余序列并保持活性,这与体外实验的结果并不一样。研究人员随即尝试把两个靶向不同位点的sgRNA直接串联在一起,发现两者均能有效地在目标位点产生突变,并且这两个sgRNA的排列顺序不存在位置效应。在这个TRBO表达系统中,甚至可以把GFP的编码序列设置在sgRNA的5′或3′端,同时执行GFP表达和sgRNA介导靶位点突变的功能。在这项研究中,尽管在植物体内sgRNA的5′和3′端能容忍多余序列这一现象已经证实,但其具体机制并未明确,作者推测了3个可能的原因:(1)在植物体内Cas9蛋白结合sgRNA时可以容忍其两端的冗余序列;(2)在植物体内Cas9蛋白可以切除sgRNA两端的多余序列;(3)烟草自身的一些蛋白能够帮助切除sgRNA两端的多余序列。
2017年,来自日本的Endo研究组通过一系列的体内和体外实验进一步证实Cas9蛋白和植物内源的RNA酶,如RNA酶Ⅲ或RNA酶T1,对于带多余序列的sgRNA的成熟是必需的[27]。在这项研究中,作者提出了以下假设的模型:在植物体内,Cas9蛋白可以结合包含sgRNA的转录产物,然后植物内源的RNA酶可以帮助切除sgRNA两端的多余序列进而形成成熟的Cas9-sgRNA复合体。当然,具体的作用流程和机理仍有待进一步研究。利用这一机制,研究人员简化了多基因敲除的载体模型:把Cas9的编码序列和一到多个sgRNA直接串连在一起通过一个pol Ⅱ启动子驱动,sgRNA之间只需一个短的linker序列(12 bp)作间隔。通过对水稻两个内源基因进行双基因敲除实验表明,该系统的基因敲除效率与包含核酶元件的多敲系统相当。
朱健康研究组进一步提出了植物多基因编辑简化单转录元件系统(simplified single transcriptional unit, SSTU)的概念[28](图 5)。在该系统的设计中,不论是利用Cas9还是Cpf1做多基因敲除,均可以把Cas9或Cpf1的编码序列和一个或多个sgRNA直接串连在一起,由一个pol Ⅱ启动子驱动形成一个表达盒。Cas9系统里的各个sgRNA之间由一个很短的linker序列(可以只有几个碱基)隔开,而Cpf1系统里的各个guide之间则由其独有的DR序列作间隔。为验证这一概念,研究人员分别构建了FnCpf1和LbCpf1的SSTU系统,同时靶向水稻内源的8个和9个基因位点,同时,还构建了基于Cas9的SSTU系统对3个水稻内源基因进行多位点敲除。水稻稳定转化结果表明,SSTU系统的多基因编辑效率总体上与经典的MCTU或TCTU系统的效率相当。

A/NOS: poly A或NOS等终止子。
图 5 SSTU系统
在目前主要利用Cas9和Cpf1作为基因编辑工具的技术条件下,SSTU系统已经把植物多基因编辑体系简化到了极致。目前该体系,特别是基于Cas9的SSTU系统的主要问题在于,尽管Endo研究组的实验已经证明Cas9蛋白和植物内源的RNA酶对于系统里sgRNA的成熟是必需的,但是具体的切割分离机理仍不明确。与Cpf1对其DR序列的特异识别切割机制不同,Cas9和植物内源的RNA酶对包括linker在内的sgRNA前体序列的处理可能是一种随机的非特异性切割。截至目前,基于Cas9的SSTU系统仅在拟南芥、水稻和烟草中对3个以内的靶位点进行过多基因敲除实验,对更多靶点的测试及在其他物种上的优化应用有待进一步深入。
6 问题及展望
在短短3~4年时间里,植物多基因编辑载体系统就伴随着Cas9和Cpf1等基因编辑工具的迅猛发展而经历了四代的进化,多个sgRNA模块从连续组装到利用RNA自剪切元件进行串联再到单转录系统的一体化应用,总体上由繁至简不断优化,载体构建越来越简单方便,必将加速植物多基因突变体的获得,打破功能冗余,进一步促进植物基因家族或通路的机理机制研究以及推动作物的改良。
目前植物多基因编辑系统仍然存在以下几个问题:(1)尽管已经在水稻、拟南芥、烟草、番茄等多种植物上进行过多基因编辑,但主要应用的还是第一代的MCTU体系,简化版多基因编辑系统的使用还少有报道,还需要在更多植物上进行适用性及效率的评价。(2)目前的多基因编辑体系主要依赖SpCas9和Cpf1构建,靶位点的设计受到这些基因编辑工具酶PAM序列(SpCas9为NGG,Cpf1为TTTV,V = A/C/G)的限制,所以,需要更多的基因编辑工具酶来做多基因编辑以提供更灵活的靶位点选择。2018年报道的XCas9就是一个很好的候选工具,它从SpCas9人工进化而来,可以识别NG、GAA和GAT的PAM序列,并且特异性更高[29]。而LbCpf1的变体LbCpf1 (RR)可识别TYCV (Y = C/T, V = A/C/G)的PAM序列,并且已经在水稻的基因敲除上得到验证[30],也可以用来建立多基因编辑系统。(3)面向多个靶位点的系统设计及靶向效率有待进一步提高。在进行多基因编辑时,尽管多数位点靶向效率较高,但还是会经常出现个别靶点突变率低下,甚至没有突变的情况,导致一次性多基因全部敲除的努力前功尽弃。在多基因编辑系统里,sgRNA阵列内是否存在RNA结构上的相互拮抗或干扰而导致其中一些sgRNA单元不能完全分离或被过度切割,最终导致个别靶点效率低下,目前尚没有明确的研究。在植物基因编辑领域,针对单个位点的sgRNA设计预测软件已经不断完善,但是面向多个靶位点进行系统设计及模拟分析的程序还鲜有报道。(4)除了sgRNA本身可能会造成脱靶之外,多基因编辑系统还可能会带来额外的脱靶效应和细胞毒性风险。为了系统的简化而添加的各种RNA加工剪切工具所识别切割的序列一般都很短,例如其中一些Hammerhead核酶的识别位点仅有15 bp,而以Cpf1构建的多基因编辑系统里,能被Cpf1蛋白自身识别切割的DR序列也仅有20 bp左右。尽管Hammerhead核酶和Cpf1均能通过序列及结构的双重特异性对其识别位点进行切割,但仍有可能对细胞体内相同或类似的转录序列进行误切从而产生额外的脱靶或毒性效应,需要在实际应用中深入观察分析。总体而言,在经历过多代简化之后,植物多基因编辑系统未来的发展方向将是更广谱、更高效和更安全。
来源:frontiersin 植物科学最前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247491686&idx=1&sn=7770dbfdf659876a9259139031b16cb4&chksm=e8bd9278dfca1b6e4ccdf48bfb57f80eac9dc8f4451c4548ce7ca6f3c5a1dcf3f86203c4f6f4&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
基因编辑 启动子 基因工程 基因敲除 基因位点 rna干扰 转录起始位点 朱健康

基因编辑“剪”出肉多黄颡鱼,国际首次敲除水产养殖鱼类基因

材料基因工程加速新材料研发
大师谈动画版:基因工程
通过目标启动子基因编辑及病原菌群体监控实现水稻白叶枯病持久广谱抗性
增强子选择启动子的新模型
SpliceR-利用碱基编辑器靶向剪接位点比iSTOP更有效的基因敲除策略!

神经领域中「特异性启动子AAV」的妙用

刘耀光院士团队开发精短型小核RNA启动子,为植物多基因编辑提供有力工具

全面分析拟南芥、玉米和高粱核心启动子,助力设计新启动子和作物改良

中国科学家培育出基因编辑瘦肉猪:脂肪少24%