科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-06-22
来源:BioArt
动物基因编辑技术是指对动物基因组特定DNA片段进行敲除、插入以及定点突变,使基因编辑动物的生物性状定向改变且可稳定遗传给后代的技术。早期的基因编辑技术包括锌指核酸酶(Zinc-finger nucleases,ZFNs)技术和转录激活因子样效应因子核酸酶(Transcription activator-like effector nucleases,TALENs)技术,但是存在制备成本高且成功率低、操作复杂等缺点,限制了基因编辑技术的发展。2012年,CRISPR/Cas9系统基因定点编辑技术的研发将基因编辑带入了一个新的时代,它只需要改变很短的sgRNA序列就可引导Cas9蛋白进行定点编辑,极大地降低了成本以及简化了构建过程,其高效性、高性价比以及良好的编辑效果受到科研人员的青睐并为人类疾病的治疗打开了新视界【1】。
尽管直接靶向DNA有其深远的临床意义,但是用DNA编辑技术靶向只有微小变化的基因来治疗相应疾病仍有很大困难,而且,其潜在的副作用以及基因永久改变的风险并不适合某些疾病。因此科学家们逐渐发现,靶向RNA水平的信息为基础研究和临床治疗发展带来了更多机会,包括:可以在副作用出现时及时停止治疗,可以靶向一些DNA水平改变存在风险的基因,可以非永久性地去控制基因表达等等。随着研究的深入,对RNA的调控过程除了通过抑制或增强改变其表达外,RNA表观转录组水平的调控也日益受到重视,其机制包括编辑、降解、转运和翻译等【2,3】。
类似于DNA编辑的CRISPR/Cas9系统,靶向RNA的CRISPR/Cas13系统的发现开启了在RNA水平研究、诊疗的新时代。活化的Cas13具有独特的核酸酶活性,可特异性地切割靶向RNA,同时非特异性地切割周围环境中的RNA,实现体外核酸检测。通过活性位点突变可产生无核酸酶活性但可与RNA结合的dead Cas13(dCas13),与其他功能性蛋白质进行融合可进一步扩大dCas13的应用范围【4】,比如通过传递ADAR A-to-I编辑酶对RNA进行编辑【5】,或者通过hnRNPa1靶向目标转录本来调节剪切【6】,也可以通过传递m6A reader蛋白至转录本特定位点来调控RNA【7】。
经过Cas9和Cas13系统对于DNA和RNA的研究是革命性的,但是其仍存在两个明显的局限性:其一是Cas蛋白巨大的分子量,使得病毒包装以及蛋白转运变得困难;其二是Cas蛋白来源于细菌,使得长期临床应用时易产生免疫原性。这两点使得基因编辑的基础研究应用和临床治疗面临着困难。
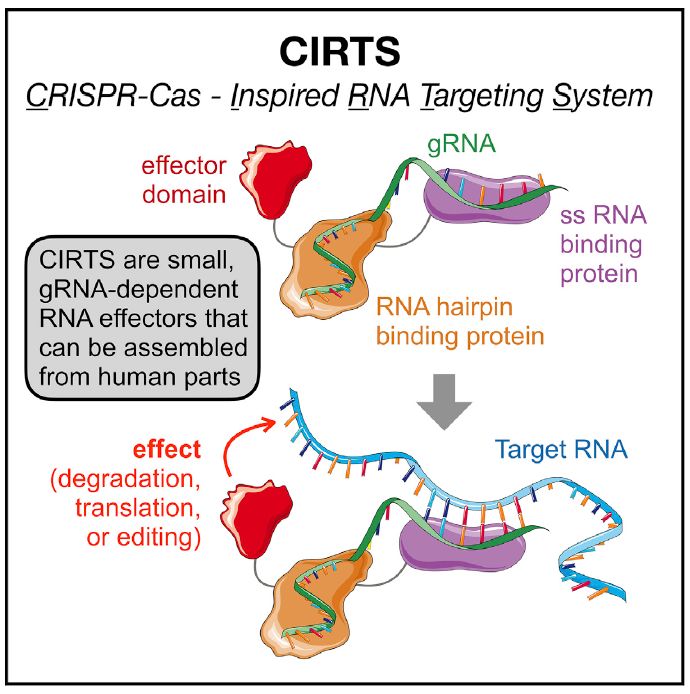
为了解决上述两个问题,2019年6月20日,来自美国芝加哥大学的Bryan C. Dickinson研究团队在Cell上发表题为Programmable RNA-Guided RNA Effector Proteins Built from Human Parts的文章,呈现了一个CIRTS系统(CRISPR-Cas inspired RNA targeting system),它是一个完全由人类蛋白成分合成的、可设计的、模块化的、RNA指导的RNA靶向效应器,克服了CRISPR-Cas系统的大分子量和免疫原性的限制性,为RNA调控的研究和利用提供了新的工具,为未来临床治疗提供了新机遇。

基于目前Cas13系统的特点,本文作者推断一个最小限度的可设计的RNA靶向体系需要4个组成部分:1)一个RNA发夹结合蛋白(RNA hairpin binding protein),作为该系统的核心,在指导RNA(gRNA)的引导下,其可以选择性并高度亲和地与特异性的RNA结构结合;2)一个gRNA,它的结构可以与设计好的发夹结合蛋白相互作用,同时它的序列与靶RNA互补;3)一个带电荷的蛋白(ssRNA binding protein),可以与gRNA序列非特异性的结合,从而在靶向事件发生之前稳定并保护gRNA;4)一个效应蛋白(effector protein),比如核糖核酸酶或者表观转录调节子,可以对目标RNA起特定作用。尽管Cas13以一个单一蛋白结构域包含了所有这些功能成分,但是本文研究者期望设计一个系统,联合多种蛋白结构域,每一种都可以展现其中一个功能,即本文所展现的CRISPR-Cas激发的RNA靶向系统——CIRTS(图1)。

图1
本文设计的第一代系统——CIRTS1,按照上述原则,是一个由ORF5-TBP6.7-Pin nuclease结构域与相应的gRNA组成的复合体,体外和细胞内实验均证实其降解目的RNA的能力与Cas13系统相差不大。进一步的,研究人员开始探索CIRTS体系中四个组分(gRNA、发夹结合结构域、非特异性RNA结合结构域和效应蛋白)的改变是否可以为CIRTS带来不同的功能。
将CIRTS1的效应蛋白由Pin nuclease变成YTHDF1(细胞质中促进翻译的m6A reader蛋白)后,目标RNA的水平没有变化,但是相应的蛋白表达增加;而将YTHDF1换成YTHDF2(促进RNA降解的m6A reader蛋白)时,目标转录本的RNA及相应的蛋白水平均下降。由此证明CIRTS可以和dCas13体系一样传递RNA表观转录调节子“reader”蛋白。而将效应蛋白换成其他功能的蛋白如hADAR2(作用于RNA的人腺苷脱氨酶2,可用于RNA编辑),可以起到RNA编辑的作用。与此同时,研究人员也发现,人源蛋白组蛋白茎环结合蛋白(SLBP)可以替代CIRTS1的发夹结合结构域TBP6.7,产生同等效应,而人源阳离子蛋白肝素结合EGF样生长因子(HBEGF)和β-defensin 3也可以替代ORF5作为非特异性RNA结合结构域起相同作用,由此证实只要符合原则,多种人源蛋白都可以作为CIRTS的发夹结合结构域和非特异性RNA结合结构域起作用。综上证明CIRTS设计的模块化使得CIRTS可以具有多样功能。
进一步地,通过设计针对不同内源性RNA的sgRNA,实验证实,CIRTS可以以一种依赖于gRNA的方式对内源性转录本起作用,可以通过激活核酸酶对靶标的活性或者触发内源性表观转录调控通路来介导降解作用,也可以激活翻译过程而增加目标转录本来源的蛋白表达量。此外,研究人员也已证实,一个CIRTS可以同时靶向多个转录本,同时,CITRS也可以同时传递多种调节效应蛋白,例如靶向降解一个转录本的同时激活另一个转录本的翻译。
除了CIRTS的组成完全是人源性的这个优点外,其另一个关键的优势在于它的分子量小,这就使得CIRTS的病毒包装和传递更有效。腺相关病毒(AAV)由于其血清可用范围广、免疫应激水平低、基因组插入风险小等优点,使其成为向多种细胞转基因以及基因治疗的有效运载工具,但是AAV的包装能力仅限于4.7 kb,这就使得Cas13这样的大分子蛋白的包装和转运受限,而本文实验发现,整个CIRTS系统只有2.7 kb(图2),当用AAV传递CIRTS至细胞内时,其对靶标的降解作用仍旧有效,从而证明CIRTS的病毒传递保持了它的功能,为临床应用提供了有效方法。
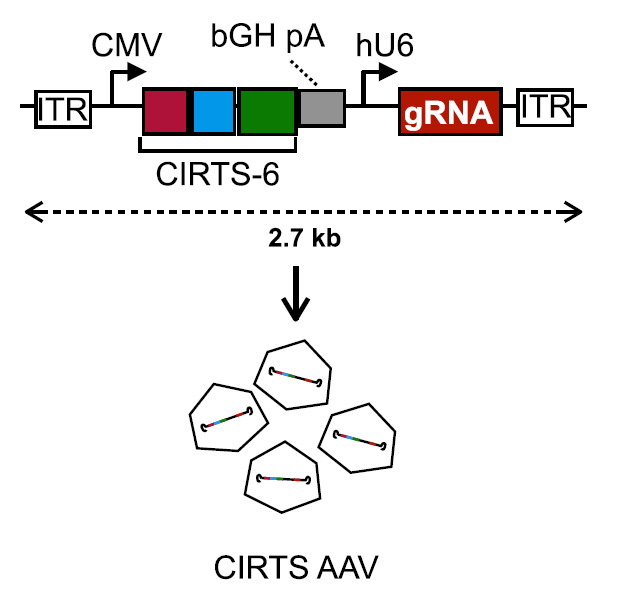
图2
综上所述,本文呈现了一个新型的、多功能的、可设计的、可编程RNA效应蛋白系统——CIRTS,其组成完全来源于人类蛋白成分,因而可以避免机体免疫反应,为持续基因治疗打开了大门;同时由于其尺寸足够小,可以用AAV有效传递;此外其可以在活细胞内靶向内源性RNA,核酸酶、表观转录调节子以及编辑器均可被CIRTS所传递,在多维度上实现对转录组的控制。这个新型RNA编辑工具的诞生,为基因编辑的研究以及临床应用带来了无限潜能!
原文链接:
https://doi.org/10.1016/j.cell.2019.05.049
参考文献
1. Sander J D, Joung J K. CRISPR‐Cas systems for editing, regulating and targeting genomes[J]. Nat Biotechnol, 2014, 32(4): 347‐55.
2. Nishikura, K. (2010). Functions and regulation of RNA editing by ADAR deaminases. Annu. Rev. Biochem. 79, 321–349.
3. Roundtree, I.A., Evans, M.E., Pan, T., and He, C. (2017). Dynamic RNA modifications in gene expression regulation. Cell 169, 1187–1200.
4. 彭利君,许红攀,张葵. 核酸检测和基因编辑的新工具:CRISPR/Cas13系统[J]. 临床检验杂志. 2019,37(05),380-382.
5. Cox, D.B.T., Gootenberg, J.S., Abudayyeh, O.O., Franklin, B., Kellner, M.J., Joung, J., and Zhang, F. (2017). RNA editing with CRISPR-Cas13. Science 358, 1019–1027.
6. Konermann, S., Lotfy, P., Brideau, N.J., Oki, J., Shokhirev, M.N., and Hsu, P.D. (2018). Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell 173, 665–676.e14.
7. Rauch, S., He, C., and Dickinson, B.C. (2018). Targeted m6A reader proteins to study epitranscriptomic regulation of single rnas. J. Am. Chem. Soc. 140, 11974–11981.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652471574&idx=2&sn=61bd5d8568749598d438c0547c55055f&chksm=84e212a2b3959bb4a0172af0b89a1ca89c115a3e25fef1944499153167e27573c2bdaf28ab9e&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

Cell :噬菌体抗菌疗法开发
Cell Stem Cell | m6A阅读蛋白YTHDF2在白血病中的功能研究

Cell Stem Cell:自然界的奇葩蛋白,驯服基因组的“跳跃”序列

中文解读 | 基底膜在血脑屏障发育及维护屏障功能中的作用

活动预告丨“CAAI进校园”系列活动走进电子科技大学

Cell Stem Cell | 骨骼肌干细胞如何应对环境污染物刺激?
Cell Stem Cell | 夏云组利用类器官揭示肾脏发育中的谱系关系


Cell Stem Cell :陈梦飞博士等揭示慢性炎症中嗅觉干细胞的角色转换
Cell:肺病为什么咳嗽?

Cell Stem Cell:中南大学林戈等揭示不同亲本对人类植入前发育的影响