科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-07-16
来源:X一MOL资讯
幽门螺杆菌(Helicobacter pylori, H. pylori)是一种寄生在人胃部的微厌氧型革兰氏阴性菌。全球约有半数的人口被其感染,它是胃炎、胃溃疡、胃腺瘤和胃淋巴瘤等胃部疾病发病的关键因素之一。幽门螺杆菌能产生一系列的疾病相关的毒力因子来帮助其在胃部增殖。幽门螺杆菌产生的高温需求蛋白A(HpHtrA)就是与细菌感染密切相关的毒力因子之一。
HpHtrA以及它的同源蛋白(HtrA)广泛的分布于原核和真核生物界,它们属于一个进化上高度保守的热激丝氨酸蛋白酶和分子伴侣家族。在绝大多数革兰氏阴性菌中,HtrA蛋白处于胞间质部位。由于其具有丝氨酸蛋白酶和分子伴侣的功能,HtrA能够通过降解或者修复周质空间中折叠不正确的蛋白来控制蛋白的质量,维持机体蛋白内稳态。然而研究发现HpHtrA则会被幽门螺旋杆菌分泌到细菌外,HpHtrA在生理功能上与其他同原蛋白也有着显著的差异。HpHtrA能够切割宿主细胞表面粘附蛋白(E-cadherin)的胞外结构域,从而打开胃壁上皮细胞间的联结,使得幽门螺杆菌能过穿过胃上皮细胞而感染胃部。因此,HpHtrA蛋白是幽门螺杆菌感染哺乳动物胃部的重要致病因子之一,有效抑制HpHtrA蛋白的活性将极大的削弱幽门螺杆菌侵染人体的能力。因此HpHtrA是一个潜在的抑制幽门螺杆菌感染的药物靶标。
基于此,中山大学化学学院夏炜副教授研究组利用蛋白质晶体学手段解析了HpHtrA蛋白的三维结构,发现了其N端结构域参与的特殊的Domain swapping互作模式,阐明了N端结构域影响HpHtrA聚集状态及其酶活性的分子机制,并基于结构模拟出了HpHtrA识别并切割底物E-cadherin的可能的机制。本研究为后续基于结构设计研发靶向HpHtrA的抗菌药物提供了基础。
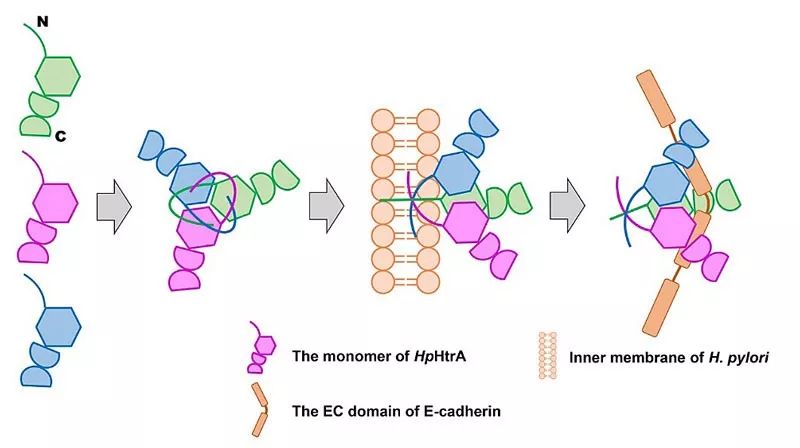
图1. HpHtrA三聚体组装及识别底物E-cadherin机制示意图
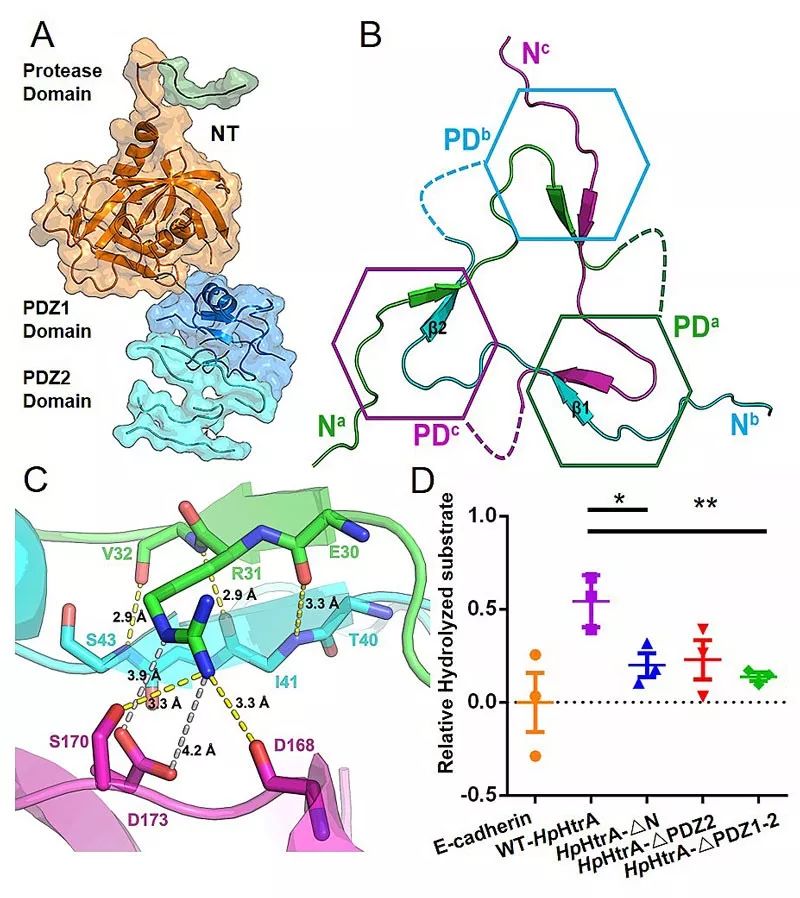
图2. N端结构域对HpHtrA的组装和功能发挥的影响。(A)HpHtrA单体晶体结构。(B)N端结构域Domain swapping示意图。(C)N端结构域具体相互作用示意图。(D)N端结构域对活性的影响。
这一研究成果最近发表在The Journal of Biological Chemistry 上。中山大学化学学院博士后张哲民和硕士研究生黄祺为论文共同第一作者,通讯作者为夏炜副教授。该项目获得国家自然科学基金、教育部创新团队、广州市科技创计划项目的支持。
来源:X-molNews X一MOL资讯
原文链接:http://mp.weixin.qq.com/s?__biz=MzAwOTExNzg4Nw==&mid=2657615531&idx=6&sn=75f07a6ba6a0af8d1e667809d0d93341&chksm=80f7d77bb7805e6d5b821686cf651c3fb8424ee6fe7b0a689dc1348c2d950b3067289a660014&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
【科普】幽门螺杆菌的自述

远离幽门螺杆菌,科普小课堂
幽门螺杆菌的“双面人生”
牙膏可以刷去幽门螺杆菌吗
幽门螺杆菌的传染途径有哪些?
幽门螺杆菌检测
吃益生菌,可以消灭幽门螺杆菌?
幽门螺杆菌的传染途径有哪些?
骇人听闻的幽门螺杆菌
认识幽门螺杆菌