科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-12-05
来源:植物生物学
摘 要:含有核苷酸结合结构域和富含亮氨酸重复序列的识别受体(NLRs)用于识别植物细胞内环境的病原感染,介导多种免疫机制。NLR介导的多种免疫机制在多个物种间独立产生,包括NLRs传感器和辅助因子。NLR亚家族中直接和间接识别机制独立出现,小RNA调控NLRs网络。了解NLRs的进化史有助于阐明病原体识别的起源和植物免疫系统的制约因素。设计抗病性的尝试很少,也很少得到进化知识的支持。这篇综述讨论了NLRs的进化史,概述了抗病基因工程的进展,并提出了利用进化知识推进未来对新的病原识别能力的研究。
1 前言
1.1 植物细胞的免疫感知
植物中存在大量质膜受体(模式识别受体)感知细胞外病原入侵,感知信号包括微生物相关分子模式或分子活动(57),免疫反应包括活性氧(ROS)积累、胞内钙离子浓度增加、丝裂原活化蛋白(MAP)激活、防御基因表达等(18)。目前,模式识别受体和感知信号,及其激活通路是研究热点(37; 78)。
病原体分泌到植物细胞的分子称为效应子,其功能是促进病原入侵,抑制植物免疫。细胞质免疫受体可识别病原体分泌到植物细胞的分子以及植物成分的酶修饰,称为含有核苷酸结合结构域和富含亮氨酸重复序列的识别受体(NLRs)。NLRs对效应子的识别会诱导一些类似质膜受体的反应,包括ROS积累、MAP激活、防御基因表达等(45)。此外,NLRs的激活会导致局部细胞死亡,称为过敏反应(HR)。
1.2 NLRs是细胞内免疫受体
NLRs由一个核苷酸结合结构域(NB-ARC)和C端富亮氨酸重复序列(LRRs)组成,大部分含有Toll/白介素受体结构域(TIR)、卷曲螺旋域(CC)、或类似于抗白粉病结构域(RPW8)(113)。NLRs根据N端结构域和NB-ARC进化史分为三类:TIR-NLRs(TNLs)、CC-NLRs(CNLs)、RPW8-NLRs(RNLs)(图1a)(113)。部分NLRs没有所有结构域,如一种细菌Ⅲ型效应蛋白的应答因子RBA1仅含有TIR结构域(96),且仅含有TIR或RPW8结构域的蛋白就能诱发病原抗性(94; 135; 145)。NLRs可以直接结合并识别效应子(46; 62),或间接地识别植物成分修饰(129; 150)。植物也有整合结构域(IDs)的免疫受体,它们模仿病原体靶标,并被效应子激活(28; 70; 105)。
NLRs可分为两个功能类群:参与病原识别的直接/间接传感器NLRs、参与免疫激活的辅助NLRs(65)。NLRs是由一个传感器和一个辅助因子组成,并有遗传联系。辅助NLRs从一一对应进化成复杂的NLRs网络,如菊类植物NRC基因家族(140-142)。在传感器NLRs下游起作用的RNLs辅助因子包括ADR1和NRG1,它们对抗性和细胞死亡至关重要(25; 71; 102; 143)。
1.3 NLRs免疫激活机制
传感器NLRs及其辅助因子的免疫激活分子机制尚不清楚。而保守的结构域和进化树表明NLRs激活的关键步骤是ADP转化为ATP后NB-ARC结构域的多聚化。因此NLRs被称为免疫信号的分子开关(123),ADP结合态是关闭状态,LRR与NB-ARC结构域相结合,使NLRs处于非活动状态(124)。ATP结合态是开启状态,NLRs被激活。拟南芥ZAR1是一种间接感应NLRs,它的低温电子显微镜结构证明这两种状态,并存在第三种中间状态(133; 134)。ADP结合态属于单体,在LRR和NB-ARC(螺旋结构域HD1和有翼螺旋区WHD)之间有多个分子内接触点(134)。效应子识别下LRR诱导改变蛋白质的构象,从而激活NLRs。ATP结合态属于寡聚体,诱导形成一种轮状五聚体,称为抗性小体(133),这与哺乳动物Apaf-1活性状态的凋亡小体(151)和NLRs激活的炎症小体(126)相似。ZAR1的CC通过形成α-螺旋体(133)促进聚集。
配体结合/修饰如何影响NB-ARC结构域状态有待研究。有一种可能性是效应子结合导致NLR分子构象变化,使NB-ARC结构域暴露,促进ADP转化为ATP(87; 125; 137)。ZAR1中宿主蛋白激酶PBL2与ZAR1/RKS1复合物结合导致NB-ARC旋转并丧失ADP亲和力(134)。然而,在另一个基于平衡的分子开关模型中,病原效应子对NLRs的ADP结合态没有显著的结合亲和力(15)。在该模型中,NLRs在开启和关闭状态之间构成循环,病原体效应子稳定了开启状态,使平衡向开启状态转变,导致防御反应的激活(15)。这两种模型可能共存,只是对应不同的NLRs,使免疫反应更强大,还可以适应不同的效应子结合亲和性。
目前还没有TNL配合物结构的报道,基于NB-ARC结构域的低聚化,可以推测它是类似的轮状结构。TNLs可形成低聚体,并且TIR结构域可以跨多个表面自我结合(16; 138; 148)。活性ZAR1抗性小体通过N端α-螺旋与膜结合,而活性TNL复合物可能通过TIR结构域对NAD+的酶解发出信号(59; 131)。许多植物和哺乳动物TIRs都能将NAD+裂解成烟酰胺和环ADP核糖(cADPR),这两种体系的酶活性都需要自我结合并依赖于保守的起催化作用的谷氨酸(59; 131)。
NLRs的激活诱导下游反应,最终导致局部细胞死亡。研究发现所有TNLs都通过编码类脂肪酶的EDS1和PAD4基因发出信号(50),但TIR结构域的酶解产物如何影响下游反应尚不清楚。大量研究阐明RNLs在传感器NLRs下游发挥的作用(25; 71; 102; 143)。NRG1与EDS1/衰老相关基因SAG101互作,启动拟南芥HR反应(102)。NRG1的RPW8结构域可破坏膜完整性(11)。一些CNLs基因的作用依赖于NDR1基因(1; 27),一种质膜蛋白。由于活性ZAR1抗性小体与膜相关,因此NDR1可能具有成孔活性。
2 NLRs的进化基因组
2.1 跨被子植物的NLRs进化
对100多个植物基因组序列分析发现单个物种或植物家族中不明显的NLRs进化模式(54; 113; 149)。根据NB-ARC系统发育,TNLs、CNLs和RNLs形成三个单系群,具有特异的N端结构域,TIR、CC和CCR结构域与祖先NB-ARC结构域可能存在融合(图1a)。三种NLR类型的进化分支都出现了无油樟属(Amborella)植物谱系,表明这类植物的进化较古老(113)。22个代表性被子植物NLRs的祖先重建表明每个基生植物约有23组NLRs(90; 113)。75个植物基因组中的20571个NLRs分析表明,311个NLRs家族中只有38个在单子叶和双子叶中保守存在,其他NLRs家族在植物谱系中特异存在(149)。
RNLs、TNLs和CNLs的拷贝数因植物而异(图1a)。除裸子植物外(144),RNLs通常以低拷贝数存在(54; 70; 113; 149)。RNLs具有明显的内含子保守性,无油樟属(Amborella)和双子叶植物有4个内含子,单子叶植物有3个内含子(第2个内含子丢失)(90; 113)。NRG和ADR的分化发生在被子植物分化前,保守存在于有花植物的同一染色体上(113)。而NRG基因在几种植物谱系中丢失(113)。
TNLs有两个分支,即TIR1和TIR2(105)。TIR2 NLRs存在于单子叶植物中(94; 105),TIR1 NLRs存在许多双子叶植物中,但在一些物种中缺失(113)。所有开花植物中,TNLs表现明显的内含子/外显子连接模式:第一个内含子将TIR与NB-AR分隔,第二个内含子将NB-ARC与LRRs分隔,第三个内含子将第一个LRR与蛋白质其余部分分隔(90; 113)。CNLs不存在保守的内含子(90; 113),祖先CNLs无内含子,因此CNLs中的内含子可能是后期进化而成(113)。
根据进化中保留的氨基酸基序,CNLs被分为至少4个类群(139)。单子叶和双子叶植物均包含EDVID基序,如拟南芥的ZAR1和RPM1蛋白,马铃薯的Rx蛋白,天竺葵科的Sr33/MLA蛋白(139)。一些CNLs在第一个α螺旋中含有功能保守的蛋氨酸、丙氨酸、天冬氨酸、丙氨酸(MADA)基序(2),该基序寡聚后重新排列并介导膜定位(133)。
NLRs的进化历史背景下(图1a),很明显传感器和辅助因子进化了多次。NRC辅助因子分支出现在1亿年前石竹目和菊类植物分化前,并在茄科中分化(140)。RNL辅助因子分支更古老,可追溯到裸子植物和被子植物分化前(113)。功能相似的NLRs,如RRS1/RPS4和RGA4/RGA5、RPS2和RPM1保卫着相同的植物蛋白RIN4,但它们在单子叶和双子叶植物分化前就发生了分化,这表明它们的功能可能是独立进化而来的。同样,编码识别同一病原体的基因(如RPP13和RPP1)也没有聚类,这表明NLRs之间的功能相似性并不一定反映它们的进化关系。
2.2 特定谱系分化枝的扩张和收缩
植物基因组NLRs的数量变化相差约100倍,从只存在几十个NLRs的木瓜、猕猴桃、黄瓜、西瓜(8; 54; 76; 79; 149)到存在几千个的六倍体小麦(4)。即使密切相关的物种也存在NLRs扩张和收缩(4; 8; 9; 55; 56; 63; 79; 83; 118; 149)。虽然推动NLRs扩张和收缩的选择机制尚不清楚,但它可以反映植物的生活方式,并受环境选择压力的影响。NLRs扩张和收缩影响的植物的生活方式包括水生环境(9),雌雄异体(猕猴桃、木瓜)或单独的雄性和雌性花(玉米、黄瓜)(8),群体遗传学表明环境压力使单一物种存在NLRs多样性,如适应病原的野生番茄(119; 120)和拟南芥(128),存在稳定的多态性NLRs防御机制(66)。
如何在较短的进化时间内实现NLRs的扩张或收缩?茄科作物的CNL扩张归因于长末端重复(LTR)逆转录转座子的活性(67)。NLRs本身的重复也有助于进化,促进基因重组(92; 111)或局部复制(72)。
2.3 NLRs等位基因多样性和新基因融合
NLRs基因的扩展和收缩在近缘物种中可观察到。然而,基因复制并不足以产生新的病原体识别蛋白。出生-死亡模型(91)中提出的NLRs快速进化是适用的。目前的基因组数据集表明NLRs多样化,通过基因内和基因间重组和转化产生嵌合LRRs(111),以及LRRs的点突变(82; 88)。这类NLRs能够识别高度变异甚至结构不相关的效应子,通过直接结合机制抵抗多种病原体的耐药性(17; 34; 82; 117)。具有高变异等位基因的NLRs出现在TNL和CNL分枝中,而并非单系的。
携带ID(Intergrated domain)的NLRs具有新的识别特性(28; 70; 105)。禾本科存在一个专门的NLR-ID分支(MIC1),它的ID持续变化,促进新ID传感器产生(10; 121)。NLR-ID显示了重复和染色体内易位,但结构域整合的确切基因组机制仍不清楚。小麦中的NLR-ID中ID被整合到NLRs附近,可变剪接产生融合转录本(4)。
快速产生新的识别特异性的代价自身免疫(19; 32)。在拟南芥中,NLRs的几个等位基因变异与自身免疫相关(3; 31; 75),这表明NLRs和它们的靶基因可能不匹配。拟南芥NLR RPP7的等位基因变异被证明与不相容的RPW8等位基因结合时会引起自身免疫反应(67)。推测在玉米等高度自交系作物的基因组中NLRs的低数量是由自身免疫导致NLRs失配的结果(8; 118)。
2.4 传感器NLRs及其辅助因子的功能网络
R基因和效应子的‘基因对基因’假说(51)目前演变为传感器和辅助因子的功能网络。RPS2和RPM1是传感器NLRs的典型例子,它们可以通过保护效应子靶标RIN4(7; 13; 39; 85; 86)来跟踪多个效应子。其他NLRs如ZAR1监测多个同源的辅助因子,每个辅助因子跟踪一个的效应子(12; 73; 80; 108; 112; 132)。具有WRKY结构域的RRS1等位基因可以检测至少两个效应子AvrRps4和PopP2(42; 84; 106; 107),并且RRS1/RPS4可以对三种病原产生抗性(95)。虽然一个NLR可以识别多个效应子,但是独立进化的NLRs也可以识别同一效应子。大豆和拟南芥的RIN4分别受NLRs Rpg1b/Rpg1r和RPM1/RPS2保护,对假单胞菌的效应子AvrRpm1和AvrB的识别独立进化(6)。另一假单胞菌具有蛋白酶活性的效应子AvrPphB,其识别NLRs在拟南芥和大麦中进化两次,但NLRs保护同一靶标PBS1(24)。同源NLRs如MLA/Sr33/Sr50和Rx1/Gpa2,通过等位基因变异进化出对不同效应子的识别(82; 117)。这些研究表明,传感器NLRs的识别特异性不能仅基于一级序列而确定。
NLRs辅助因子可以形成自己的功能网络。在拟南芥中,TNLs和CNLs可以通过NRG1和ADR1亚支的蛋白发出信号,其中TNLs倾向于NRGs,而CNLs倾向于ADRs(25; 71; 102; 143)。NRC辅助因子分枝是一个更新的网络,与CNL传感器密切相关(140)。尽管配对的NLR基因(如RGA4/RGA5和RRS1/RPS4)的蛋白产物表现相互作用(29; 61),但目前NLRs对NRC和RNL的功能依赖仅表现在基因水平上(25; 71; 102; 140; 143)。蛋白-蛋白互作是否仅存在于配对的NLRs中,是否在辅助因子网络的形成过程中丢失,仍有待确定。
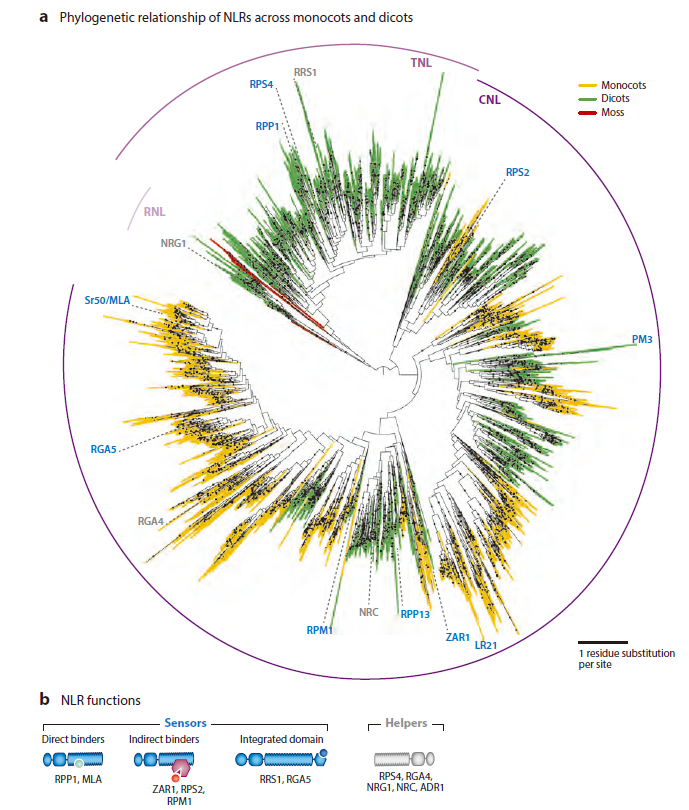
图1 NLRs在开花植物中的进化和功能。
(a)基于NB-ARC区域的7133个NLRs的系统发育来自11个双子叶植物(绿色)、7个单子叶植物(黄色)和1个苔藓植物(红色)(11),NLRs的主要类别包括RNLs(浅紫色)、TNLs(中紫色)、CNLs(深紫色),蓝色基因表传感器、灰色基因表辅助因子,单子叶植物包括水稻、高粱、小麦、斜麦、大麦、玉米、短轴草,双子叶植物包括拟南芥、蒺藜苜蓿、马铃薯、番茄、葡萄、大豆、苹果、碧桃、巨桉、野草莓、毛果杨,苔藓为小立碗藓。(b)NLR功能示意图,蓝色表传感器,灰色表辅助因子,病原效应子以圆表示,病原靶向的植物蛋白和整合结构域呈六边形,效应子识别位点呈三角形。
3、NLR生物学趋同进化的模式
3.1 NLR进化过程中,传感器对辅助因子的依赖不止一次出现
RNL分支是一个古老的、保守的辅助因子类群,在单子叶和双子叶植物均存在,而NRCs的辅助因子功能是独立产生的(图2a)。NRC可能是1亿多年前从一个茄科NLR进化而来,并演化成一个由辅助因子和传感器组成的功能性网络(140)。两个独立的TNLs和CNLs分支,如RRS1/RPS4和RGA5/RGA4,在禾本科和十字花科中细分为传感器和辅助因子(28; 95)。禾本科和十字花科编码传感器和辅助因子的基因对位点是头对头连接的,传感器常携带一个ID(图2b)(10; 95)。驱动基因头对头聚合,融合IDs,以及功能细分的辅助因子和传感器的基因组和进化机制尚不清楚。
传感器免疫受体对其他受体的依赖不是植物所特有的。在动物中,NLR家族NAIP5是一个识别鞭毛蛋白的传感器,依赖于辅助因子NLR家族的NLRC4进行响应(126)。结合配体的受体激酶FLS2和BRI1在功能上起传感器作用,依赖于与受体BAK1的物理相互作用来进行复杂的激活和下游信号传导(35; 93)。因此,配体感应受体对附加受体的需求是普遍的进化趋势。重点是要了解这样的趋势是受体启动信号级联的共同功能所施加的,还是由于受体间需要协同发挥作用。
3.2 小RNA对NLRs的调控
NLRs在mRNA和蛋白水平上都受到严格的调控(74; 115)。对植物基因组中NLRs的整体分析揭示了NLRs由miRNAs调控,并且miRNAs可由重复的NLRs产生(图2b)。一些miRNAs,如靶向CNLs的miR482/2118,很早以前就出现,这些miRNA家族在大多数植物谱系中保守存在,靶向是NB-ARC结构域的保守P-loop区域(54)。其他miRNAs出现较晚,具有谱系特异性(58; 74; 81; 99; 110; 115; 144; 147; 149)。miRNAs的不断进化与NLRs谱系特异性扩增有关,新的miRNAs可能来源于靶向NLR序列的反向重复序列(149)。
miRNAs对NLRs的调控与小RNA(sRNAs)对快速增殖的遗传元件(转座元件TEs)的调控相似。NLRs和TEs都能快速复制,表现谱系特异性的扩增和收缩,并能在基因组中跳跃。无论是新插入的TEs还是复制的基因,都受到sRNAs介导的RNA依赖的DNA甲基化,进行表观遗传标记(97; 103)。丁香假单胞菌感染的植株的sRNAs及相关表观遗传标记变化的分析显示,TEs和NLRs均在感染早期释放,并开始产生sRNAs,其中许多sRNAs靶向NLR和TE位点(23)。在感染过程中,随着RNA导向的DNA甲基化(RdDM)在基因组中积累DNA甲基化,使TEs沉默,然而许多NLRs可以继续表达,包括ADR1,尽管存在可以靶向它们的sRNAs(23)。
启动子元件插入TEs对NLRs的调控具有重要作用(图2b)。在水稻中,在NLR基因PigmS的启动子中插入微型反向重复转座因子,可以限制其在花粉中表达,并通过RdDM沉默PigmS转录(41)。PigmS蛋白是另一种NLR PigmR的显性抑制因子,PigmR提供对稻瘟病的抗性。当PigmS沉默时,PigmR蛋白在营养组织中提供抗性。而在花粉中,它被PigmS抑制,从而减少了水稻籽粒中激活免疫信号造成的产量损失(41)。sRNAs和可逆表观遗传标记对NLR的调控,可以为设计NLR启动子提供策略,以维持免疫激活和产量损失之间的平衡。
3.3 与哺乳动物的细胞内免疫的相似性
NLRs的独立进化特征不仅在植物谱系中明显,在生物界中也是明显的。在动物中,一个NB-ARC结构域同系物NACHT独立地结合LRRs和不同的信号转导结构域:CARD、BIR、PYD、DD、DED(127)。哺乳动物NLRs具有类似的细胞内监测功能,可在病原体感知和自身免疫反应中诱导细胞死亡(64)。在真菌中,含有NACHT结构域的Het-E蛋白触发非自我识别并诱导细胞死亡,称为异核体不亲和性(100)。在RNLs中,与RPW8相似的真菌HeLo结构域具有四螺旋束折叠结构,可以插入膜中形成小孔,诱导细胞死亡(38)。
植物NLRs结构生物学的最新突破是解决ZAR1及其辅助因子RKS1和PBL2激酶的活性寡聚复合体的结构(133)。ZAR1抗性小体在结构上与Apaf-1的凋亡小体(151)和哺乳动物NLRs的炎性小体(图2c)(104; 126; 133)相似。虽然炎症小体和抵抗小体之间的结构相似性很清楚,但仍有许多问题没有解答。首先,CNLs的CC结构域是否足以在细胞膜上形成小孔并导致细胞死亡,RNLs的RPW8结构域足以在膜上形成一个孔,与真菌HeLo蛋白结构相似,显然可以诱导细胞死亡。
其次,是否所有的传感器都是抗病小体,或者抗病小体的组成是否各不相同?成对的传感器和辅助因子的比例相等,还是单个传感器引发多个辅助因子的复合物组成(图2c)?比如哺乳动物NAIP5和NLRC4(126),单个NAIP5传感器被鞭毛蛋白感知激活,在构象变化后,与辅助因子NLRC4结合,诱导NLRC4- NLRC4寡聚物形成(126)。最终炎性小体通过一个NAIP5传感器和九个NLRC4形成一个开环结构。虽然ZAR1抗性小体是同质的,但不能排除其他植物NLRs可以形成类似NAIP5/NLRC4的异质蛋白复合物的可能性。CCs可以形成由复杂的相互作用网络组成的同质或异质寡聚物(30; 87; 139),这符合异质抗病小体的概念(图2c)。CNLs常形成异质寡聚物(139),而TNLs异寡聚物的报道相对较少(61; 77)。如果成对的NLR抗病小体组成新颖,并且传感器和辅助因子等比例,将是一个新发现(图3c)。
植物和哺乳动物细胞死亡程序的相似性超过抗病小体与炎性小体的结构相似性。最近关于细菌、哺乳动物和植物TIR结构域NADase活性的报道(47; 48; 59; 131)表明,细胞死亡的激活可由保守的二级信使介导。结构分析显示,哺乳动物神经细胞死亡蛋白SARM1和植物TIRs的TIR结构域有一个保守的底物结合位点(59)。与跨界TIR蛋白的共同功能一致,在烟草中SARM1的TIR结构域的表达产生了与HR难以区分的细胞死亡反应(59; 131)。SARM1和TNLs之间的主要区别是它们的遗传需求。与植物TIRs不同,SARM1能够诱导植物细胞死亡而不依赖于EDS1和NRG1(59; 131)。阐明最终导致细胞死亡反应的精确信号级联可以揭示不同物种的相似性,并阐明EDS1和NRG1的植物特异性作用。
最后,抗病小体的结构是否代表其功能活性状态?对哺乳动物NLRs的研究表明,炎性小体的形成可伴随着蛋白酶介导的半胱天冬酶结构域的释放(104)。因为它已被广泛证明在植物的氨基端截断NLRs,TIRs或CCs足以诱导细胞死亡(16; 30; 52; 69; 122; 139),可能活性抗病小体进在植物细胞中进一步修饰,诱导HR反应。
我们可以从NLR进化模式中学到什么?其中一个主要的思路是有多种方式可以进化为相同的功能,新功能也可以以多种方式设计。这些功能的设计可以通过学习NLR进化的生物学原理来指导。
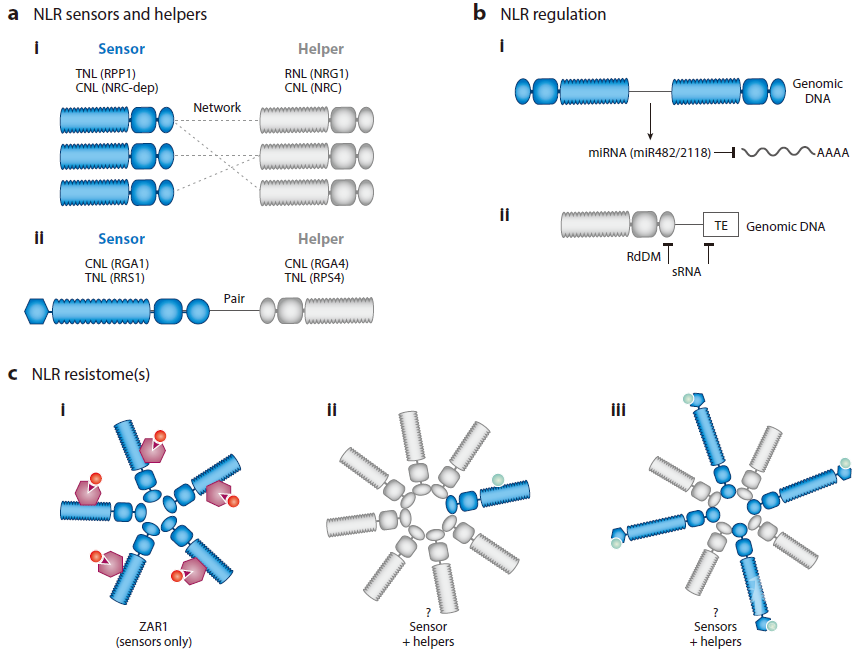
图2 NLR的生物学趋同进化模式。
(a)传感器/辅助因子网络,传感器NLRs参与识别病原体入侵,辅助因子执行免疫信号,它们在网络中相互作用。(b)NLRs受sRNAs的调控在大多数植物谱系中保守,包括源自NLR本身并参与转录沉默的microRNAs(Ⅰ),以及通过RNA介导的DNA甲基化靶向转座因子和NLR的sRNAs(Ⅱ)。(c)活化的NLRs多复合体的形成,已知的抗病小体(Ⅰ),以及假设的炎性小体(Ⅱ、Ⅲ)。
4、NLR等位基因工程的最新进展
4.1 NLR嵌合体定义效应子结合区域,并允许在等位NLRs之间识别转移
亚麻NLRs嵌合体是改变NLRs效应子识别的首次成功尝试(46)。该研究分析了亚麻抗锈病基因L的13个等位基因(L、L1-L11、LH),确定了变异区域(46)。创造L2、L6和L10嵌合体,并把它们转入亚麻,并对亚麻锈病易感性的变化进行筛选(46)。NLRs交换的区域包含880个氨基酸,包括整个LRR区域。表达这些嵌合体的转基因亚麻植株表现出与LRR相关的抗性和易感表型,也就是说含有L6-TIR-NBARC和L2-LRR的嵌合基因L6-L2表现出L2等位基因的抗性表型(46)。这一发现表明,LRR区域可以充分调控NLRs的识别特异性,直接与它们识别的效应子相互作用。自第一次在L基因进行LRR互换以来,其他几个直接结合物的识别特异性已经通过等位基因互换(114; 117)或体内相互作用(26; 34; 44; 69; 102)映射到LRR区域。
4.2 引入新的效应子底物序列到植物蛋白
NLRs可以通过监测宿主蛋白或直接结合效应子来识别效应子(图1b)。由于对效应子功能理解在不断提高,利用效应子活动设计抗性是一个很好的解决方案。Kim等(68)利用这一策略设计了RPS5的靶蛋白,使RPS5识别多个效应子。RPS5识别细菌效应子AvrPphB,AvrPphB作为一种蛋白酶作用于PBS1和相关受体类胞质激酶(RLCKs)。在被AvrPphB裂解后,RLCKs发生构象变化,被RPS5识别并导致免疫反应的激活(43)。在拟南芥感病基因型中,PBS1蛋白水解位点与另一个效应子AvrRpt2蛋白水解位点的交换显著提高了对丁香假单胞菌的抗性(68)。同样的策略也被用于将烟草腐蚀病毒的病毒蛋白酶NIa的蛋白水解位点插入到PBS1中(68)。尽管这种方法导致了NIa裂解PBS1,但它只增强了对病毒的部分抗性(68)。这一发现表明,底物裂解率和信号强度需要足够强才能达到稳定响应,对不同的效应子可能有不同的要求。因此,这种方法可能需要额外的优化和蛋白质工程来产生抗性。
4.3 引入氨基酸变化来修饰效应子识别特异性
LRR可以作为效应子结合位点并介导NLR识别特异性,促使许多人尝试随机诱变LRRs以产生新的抗病性。NLR Rx能够对携带外壳蛋白残基为T121和K127的变种马铃薯病毒x产生抗性。而具有赖氨酸和精氨酸的外壳蛋白不能被Rx识别(14)。在这项研究中,Rx通过易出错的聚合酶链反应随机突变,筛选了数千个突变体,获得识别外壳蛋白的突变体(49)。这种方法获得了几个突变的Rx蛋白,它们对携带CP-KR和CP-TK外壳蛋白的马铃薯病毒x以及另一种杨树花叶病毒表现抗性(49)。虽然这种方法扩大了对Rx的认识,但并没有产生新的抗性。
2014年发表的两项研究通过随机诱变、基因重组和定点诱变的方法,提高了野生马铃薯中NLR R3a的识别能力(33; 109)。野生型R3a识别的是疫霉菌效应子Avr3aKI,而不是等位基因变体Avr3aEM,该等位基因已成为现代疫霉病菌中普遍存在的等位基因,很可能是由于Avr3aEM避开了R3a介导的抗性从而正向选择而被保留(5; 20; 21; 130; 146)。Segretin等(109)鉴定了8个R3a单氨基酸替代突变体能够使本氏烟在AvrR3aEM响应中触发HR反应,其中6个突变位于LRR区域,1个位于NB-ARC结构域,1个位于CC结构域。其中一个突变(K920E)也在第二项研究中被证实(33)。经过几轮人工进化,Chapman等(33)发现了几个比参野生型显示出更强细胞死亡反应的突变体。然而,这些设计的NLRs,称为R3a+和R3a*,并不能增强对携带Avr3aEM的疫霉菌的抗性。
在一项后续研究中,Segretin将发现的突变转移到番茄的R3a同源基因I2上,I2通过Avr2效应子的识别对番茄镰刀菌产生抗性,并与R3a具有较高的序列相似性(98; 116)。R3a+等位基因中的两个残基(I141F和N336Y)在I2中保守(60),在I2中进行突变以查看其识别能力是否会扩大(53)。然而,这些氨基酸改变导致了I2识别或自身活性丧失(53)。假设这两个位点可能是NLR敏感化的关键,Giannakopoulou等(53)突变这两个残基,使其与所有可能的氨基酸替换,最终鉴定了I2I141N,它可以识别Avr3aKI和Avr3aEM,以及两个新的Avr2变种(53)。这些NLRs在本氏烟中的表达导致了对携带Avr3a任一疫霉菌变种产生部分抗性(53)。因此,目前为止对NLRs的随机诱变的尝试并没有导致新的抗病性的产生。
5 NLRs工程的前景与挑战
5.1 设计激活NLR的效应子靶蛋白
利用效应体活性设计新的抗病性是一个很有前景的策略,因为病原的效应子往往进化而避开与NLR的结合,同时保持其活性。有几种方法可以利用效应子活性来抵抗病原体。包括Kim等(68)采用的策略,将已知的效应子靶向蛋白水解位点转移到由植物NLR保护的植物蛋白上。或许可以设计出一种靶蛋白,它通过几个效应子的修饰后可导致NLR激活(图3a)。一个NLR能识别多少种不同类型的效应子活动?RIN4被RPS2和RPM1在内的多个NLRs保护,被多种具有不同酶活性的效应子靶向,这表明间接识别确实只能识别一种修饰,因为RPM1只有在RIN4磷酸化后才被激活,而RPS2可以识别其蛋白裂解(22)。RIN4被几个NLRs保护的事实使其成为引入更多效应子靶点的候选靶标。效应子酶活性的研究将继续扩大这一方法的潜在靶点(22)。通过选择对病原体入侵至关重要的活性效应子,可以尝试创造持久的抗性。这种方法需要对效应子活动有全面的了解。因此,了解自然效应子的靶标及其活动对这一工程至关重要。
5.2 设计新的整合结构域的NLRs
除设计靶蛋白之外,还可以使用IDs来设计NLR分支。理想情况下,可以使用现有的NLR-IDs作为平台,与效应子相互作用研究中确定的靶点进行融合(图3b)(101; 136)。目前观察到的NLR-IDs结构域自最初的融合事件以来已经协同进化了数百万年。因此,这种工程需要仔细解析协同进化区域,或鉴定仍能接受新的可变结构域融合的泛型NLR,如禾本科中发现的MIC1的NLRs(10; 121)。但还需要更好地理解NLR-ID激活机制。如,效应子对ID的修改如何改变NLR-ID的构象?NLR辅助因子在这NLR-效应子中扮演什么角色?一种观点认为辅助因子抑制了传感器NLR自身活性,而另一种观点认为辅助因子介导了免疫信号的启动(61; 89)。
在水稻的Pik等位基因序列中,整合的HMA结构域是NLR中最具多态性的区域(36),这表明它是效应子结合界面。Pik等位基因与其相应效应子的结晶证实了这一观点(89)。利用效应子结合ID这一点可以交换效应子靶向的其他已知宿主IDs结构域(图3b)。这种交换可以更好地识别效应子,通过将效应子引入现有的NLR-ID结构域以确定其靶标(图3b)。除了创造新的融合蛋白,对现有的IDs进行蛋白质工程可以改变NLR和效应子之间的结合强度,从而改善现有的识别能力。为此,从NLRs晶体结构与几个等位效应子的结合中获得的信息,提高了对结合界面以及哪些氨基酸可以提高结合亲合力的理解(40)。这些信息有助于指导蛋白质设计,以超越过去的随机诱变方法。
5.3 开发具有自然多样性LRR结合特异性的NLRs
效应子-NLR相互作用的最简单模式是通过可变LRR区域介导的直接结合,因为它涉及的遗传成分最少。因此,设计直接结合的NLRs可能是最容易设计的NLRs类型(图3c)。然而,目前缺乏这些相互作用的结构和生化数据。关键的问题包括确定LRR残基的结合特异性,产生相互作用所需的亲和力,以及引发免疫信号的结构重排。在亚麻L等位基因序列研究的基础上,可以利用自然等位基因多样性和结构建模一致地鉴定LRRs中的结合部位(46)。通过对NLRs种内自然多样性的收集,可以确定LRR中10-20个残基的多样性,这些残基是最易变的,因此很可能参与效应子结合。这些残基可以通过有针对性的工程改善其现有的结合特性或衍生新的结合特性。
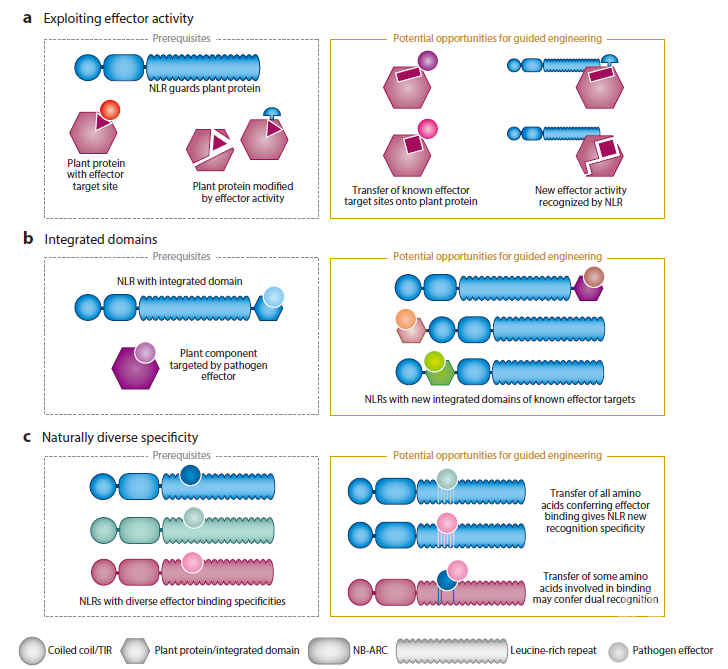
图3 新抗病性工程的进化指南。
(a)利用效应子活动设计新的识别特性;(b)在NLR-ID引入新的结构域;(c)获取自然多样性(用不同的颜色表示),以识别效应子结合残基。
6 结论与未来方向
理解NLRs的进化对于确定它们的功能、了解影响植物免疫受体库的环境压力、以及设计新的识别特异性是至关重要的。不能把一个NLR的信息应用到另一个NLR上,然而NLRs的系统发育位置能更好地表明它们是辅助因子(如RNLs)还是传感器,以及它们依赖何种类型的辅助因子(如传感器的NRC分支)。此外,IDs不仅突出了可能被病原体瞄准的植物蛋白,从而被植物免疫系统监测,而且还为效应子导向的工程方法提供了前所未有的潜力。然而,即使在一个支系中的基因之间也可能存在功能差异,就像NRG1在十字花科和茄科中的表现一样(25; 71; 102)。随着序列数据库的发展,NLR的进化存在越来越有挑战性的问题。
NLRs工程是困难的,成功的研究成果仍然有限。据我们目前对NLRs的了解,有三种主要的策略可创造新的抗病性。种群NLRs序列多样性资源为效应子结合表面的预测提供原材料。对于NLR-IDs, IDs可以作为效应子靶标的数据库。现在需要了解融合的时间以及随后与典型NLR结构域的共同进化。该信息可选择出可用于交换IDs的NLR分支。例如,来自进化中最初始分支MIC1的NLR-IDs可能拥有最大的改造潜力。不久的将来,从NLR进化中获取的信息以及合成、结构生物学的发展将使我们能够创造出更好的NLRs。
要点总结
1、核苷酸结合富亮氨酸重复受体(NLRs)在传感器和辅助因子中的亚功能化在不同物种中多次独立出现。
2、从NLR对进化而来的辅助因子比CC-NLR(RNL)的辅助因子进化更早。
3、NLRs的传感器和辅助信号元件的亚功能化让人联想到模式识别受体和协同受体之间的关系,可能是一个普遍的进化主题。NLRs中sensor和helper信号元件的亚功能化与模式识别受体的受体和共受体的关系有关,这可能是一个普遍的进化主题。
4、虽然从序列上预测NLRs的作用模式具有挑战性,但在演化支中的系统发育位置可以帮助预测其遗传需求。
5、NLRs的工程设计一直很困难,到目前为止只取得部分成功。
6、理解NLRs的演变可以为设计工程提供信息,并引导设计具有新的识别特异性的NLRs。
未来的问题
1、为什么植物会失去某些NLR枝系而扩展其他枝系?
2、进化模式在种内和种间尺度上是保守的吗?
3、人类和病原体的进化压力如何影响免疫基因的进化?在驯养系统和野生系统中是一致的吗?
4、NLRs的调控是在什么时间尺度上,通过什么方式响应随环境变化而变化的适应度?与NLR内的序列进化相比,这是以更快还是更慢的速率发生的?
5、为什么NLRs细分为辅助因子和传感器?这种关系如何运作,复合体是如何被效应子激活?
6、EDS1、PAD4和RNL辅助因子之间的功能联系是什么?导致植物超敏反应的分子机制是什么?它在植物免疫中的作用是什么?
7、整合结构域IDs的功能是什么?它们仅是病原体的诱饵,还是履行了其它功能,例如将NLR定位到正确的亚细胞区域。
8、哪些NLR-IDs可以作为新的融合蛋白的平台?哪些靶蛋白可以作为效应子靶标平台?
参考文献
1. Aarts N, Metz M, Holub E, Staskawicz BJ, Daniels MJ, Parker JE. 1998. Different requirements for EDS1 and NDR1 by disease resistance genes define at least two R gene–mediated signaling pathways in Arabidopsis. PNAS 95:10306–11
2. Adachi H, Contreras MP, Harant A, Wu CH, Derevnina L, et al. 2019. An N-terminal motif in NLR immune receptors is functionally conserved across distantly related plant species. Elife 8:e49956
3. Alcazar R, von Reth M, Bautor J, Chae E, Weigel D, et al. 2014. Analysis of a plant complex resistance gene locus underlying immune-related hybrid incompatibility and its occurrence in nature. PLoS Genetics 10:e1004848
4. Andersen EJ, Nepal MP. 2019. Diversification of disease resistance receptors by integrated domain fusions in wheat and its progenitors. bioRxiv:695148
5. Armstrong MR, Whisson SC, Pritchard L, Bos JI, Venter E, et al. 2005. An ancestral oomycete locus contains late blight avirulence gene Avr3a, encoding a protein that is recognized in the host cytoplasm. PNAS 102:7766-71
6. Ashfield T, Redditt T, Russell A, Kessens R, Rodibaugh N, et al. 2014. Evolutionary relationship of disease resistance genes in soybean and Arabidopsis specific for the Pseudomonas syringae effectors AvrB and AvrRpm1. Plant Physiology 166:235-51
7. Axtell MJ, Staskawicz BJ. 2003. Initiation of RPS2-specified disease resistance in Arabidopsis is coupled to the AvrRpt2-directed elimination of RIN4. Cell 112:369-77
8. Baggs E, Dagdas G, Krasileva KV. 2017. NLR diversity, helpers and integrated domains: making sense of the NLR IDentity. Current Opinion in Plant Biology 38:59-67
9. Baggs EL, Thanki AS, O’Grady R, Schudoma C, Haerty W, Krasileva KV. 2019. Convergent gene loss in aquatic plants predicts new components of plant immunity and drought response. bioRxiv:572560
10. Bailey PC, Schudoma C, Jackson W, Baggs E, Dagdas G, et al. 2018. Dominant integration locus drives continuous diversification of plant immune receptors with exogenous domain fusions. Genome Biology 19:23
11. Barragan CA, Wu R, Kim ST, Xi W, Habring A, et al. 2019. RPW8/HR repeats control NLR activation in Arabidopsis thaliana. PLoS Genetics 15:e1008313
12. Bastedo DP, Khan M, Martel A, Seto D, Kireeva I, et al. 2019. Perturbations of the ZED1 pseudokinase activate plant immunity. PLoS Pathogens 15:e1007900
13. Belkhadir Y, Nimchuk Z, Hubert DA, Mackey D, Dangl JL. 2004. Arabidopsis RIN4 negatively regulates disease resistance mediated by RPS2 and RPM1 downstream or independent of the NDR1 signal modulator and is not required for the virulence functions of bacterial type III effectors AvrRpt2 or AvrRpm1. Plant Cell 16:2822-35
14. Bendahmane A, Kohn BA, Dedi C, Baulcombe DC. 1995. The coat protein of potato virus X is a strain-specific elicitor of Rx1-mediated virus resistance in potato. Plant Journal 8:933-41
15. Bernoux M, Burdett H, Williams SJ, Zhang X, Chen C, et al. 2016. Comparative analysis of the flax immune receptors L6 and L7 suggests an equilibrium-based switch activation model. Plant Cell 28:146-59
16. Bernoux M, Ve T, Williams S, Warren C, Hatters D, et al. 2011. Structural and functional analysis of a plant resistance protein TIR domain reveals interfaces for self-association, signaling, and autoregulation. Cell Host Microbe 9:200-11
17. Bittner-Eddy PD, Crute IR, Holub EB, Beynon JL. 2000. RPP13 is a simple locus in Arabidopsis thaliana for alleles that specify downy mildew resistance to different avirulence determinants in Peronospora parasitica. Plant Journal 21:177-88
18. Boller T, Felix G. 2009. A renaissance of elicitors: perception of microbe-associated molecular patterns and danger signals by pattern-recognition receptors. Annual Review of Plant Biology 60:379-406
19. Bomblies K, Lempe J, Epple P, Warthmann N, Lanz C, et al. 2007. Autoimmune response as a mechanism for a Dobzhansky-Muller-type incompatibility syndrome in plants. PLoS Biology 5:e236
20. Bos JI, Chaparro-Garcia A, Quesada-Ocampo LM, McSpadden Gardener BB, Kamoun S. 2009. Distinct amino acids of the Phytophthora infestans effector AVR3a condition activation of R3a hypersensitivity and suppression of cell death. Molecular Plant-Microbe Interactions 22:269-81
21. Bos JI, Kanneganti TD, Young C, Cakir C, Huitema E, et al. 2006. The C-terminal half of Phytophthora infestans RXLR effector AVR3a is sufficient to trigger R3a-mediated hypersensitivity and suppress INF1-induced cell death in Nicotiana benthamiana. Plant Journal 48:165-76
22. Buttner D. 2016. Behind the lines-actions of bacterial type III effector proteins in plant cells. FEMS Microbiology Reviews 40:894-937
23. Cambiagno DA, Nota F, Zavallo D, Rius S, Casati P, et al. 2018. Immune receptor genes and pericentromeric transposons as targets of common epigenetic regulatory elements. Plant Journal 96:1178-90
24. Carter ME, Helm M, Chapman AVE, Wan E, Restrepo Sierra AM, et al. 2019. Convergent evolution of effector protease recognition by arabidopsis and barley. Molecular Plant-Microbe Interactions 32:550-65
25. Castel B, Ngou PM, Cevik V, Redkar A, Kim DS, et al. 2019. Diverse NLR immune receptors activate defence via the RPW8-NLR NRG1. New Phytologist 222:966-80
26. Catanzariti AM, Dodds PN, Ve T, Kobe B, Ellis JG, Staskawicz BJ. 2010. The AvrM effector from flax rust has a structured C-terminal domain and interacts directly with the M resistance protein. Molecular Plant-Microbe Interactions 23:49-57
27. Century KS, Shapiro AD, Repetti PP, Dahlbeck D, Holub E, Staskawicz BJ. 1997. NDR1, a pathogeninduced component required for Arabidopsis disease resistance. Science 278:1963–5
28. Cesari S, Bernoux M, Moncuquet P, Kroj T, Dodds PN. 2014. A novel conserved mechanism for plant NLR protein pairs: the "integrated decoy" hypothesis. Frontiers in Plant Science 5:606
29. Cesari S, Kanzaki H, Fujiwara T, Bernoux M, Chalvon V, et al. 2014. The NB-LRR proteins RGA4 and RGA5 interact functionally and physically to confer disease resistance. the EMBO Journal 33:1941-59
30. Cesari S, Moore J, Chen C, Webb D, Periyannan S, et al. 2016. Cytosolic activation of cell death and stem rust resistance by cereal MLA-family CC-NLR proteins. PNAS 113:10204-9
31. Chae E, Bomblies K, Kim ST, Karelina D, Zaidem M, et al. 2014. Species-wide genetic incompatibility analysis identifies immune genes as hot spots of deleterious epistasis. Cell 159:1341-51
32. Chae E, Tran DT, Weigel D. 2016. Cooperation and conflict in the plant immune system. PLoS Pathogens 12:e1005452
33. Chapman S, Stevens LJ, Boevink PC, Engelhardt S, Alexander CJ, et al. 2014. Detection of the virulent form of AVR3a from Phytophthora infestans following artificial evolution of potato resistance gene R3a. PLoS One 9:e110158
34. Chen J, Upadhyaya NM, Ortiz D, Sperschneider J, Li F, et al. 2017. Loss of AvrSr50 by somatic exchange in stem rust leads to virulence for Sr50 resistance in wheat. Science 358:1607-10
35. Chinchilla D, Zipfel C, Robatzek S, Kemmerling B, Nurnberger T, et al. 2007. A flagellin-induced complex of the receptor FLS2 and BAK1 initiates plant defence. Nature 448:497-500
36. Costanzo S, Jia Y. 2010. Sequence variation at the rice blast resistance gene Pi-km locus: Implications for the development of allele specific markers. Plant Science 178:523-30
37. Couto D, Zipfel C. 2016. Regulation of pattern recognition receptor signalling in plants. Nature Review Immunology 16:537-52
38. Daskalov A, Habenstein B, Sabate R, Berbon M, Martinez D, et al. 2016. Identification of a novel cell death-inducing domain reveals that fungal amyloid-controlled programmed cell death is related to necroptosis. PNAS 113:2720-5
39. Day B, Dahlbeck D, Staskawicz BJ. 2006. NDR1 interaction with RIN4 mediates the differential activation of multiple disease resistance pathways in Arabidopsis. Plant Cell 18:2782-91
40. De la Concepcion JC, Franceschetti M, Maqbool A, Saitoh H, Terauchi R, et al. 2018. Polymorphic residues in rice NLRs expand binding and response to effectors of the blast pathogen. Nature Plants 4:576-85
41. Deng Y, Zhai K, Xie Z, Yang D, Zhu X, et al. 2017. Epigenetic regulation of antagonistic receptors confers rice blast resistance with yield balance. Science 355:962-5
42. Deslandes L, Olivier J, Peeters N, Feng DX, Khounlotham M, et al. 2003. Physical interaction between RRS1-R, a protein conferring resistance to bacterial wilt, and PopP2, a type III effector targeted to the plant nucleus. PNAS 100:8024-9
43. DeYoung BJ, Qi D, Kim SH, Burke TP, Innes RW. 2012. Activation of a plant nucleotide binding-leucine rich repeat disease resistance protein by a modified self protein. Cellular Microbiology 14:1071-84
44. Dodds PN, Lawrence GJ, Catanzariti AM, Teh T, Wang CI, et al. 2006. Direct protein interaction underlies gene-for-gene specificity and coevolution of the flax resistance genes and flax rust avirulence genes. PNAS 103:8888-93
45. Dodds PN, Rathjen JP. 2010. Plant immunity: towards an integrated view of plant-pathogen interactions. Nature Review Genetics 11:539-48
46. Ellis JG, Lawrence GJ, Luck JE, Dodds PN. 1999. Identification of regions in alleles of the flax rust resistance gene L that determine differences in gene-for-gene specificity. Plant Cell 11:495-506
47. Essuman K, Summers DW, Sasaki Y, Mao X, DiAntonio A, Milbrandt J. 2017. The SARM1 Toll/Interleukin-1 receptor domain possesses intrinsic NAD(+) cleavage activity that promotes pathological axonal degeneration. Neuron 93:1334-43
48. Essuman K, Summers DW, Sasaki Y, Mao X, Yim AKY, et al. 2018. TIR domain proteins are an ancient family of NAD(+)-consuming enzymes. Current Biology 28:421-30
49. Farnham G, Baulcombe DC. 2006. Artificial evolution extends the spectrum of viruses that are targeted by a disease-resistance gene from potato. PNAS 103:18828-33
50. Feys BJ, Moisan LJ, Newman MA, Parker JE. 2001. Direct interaction between the Arabidopsis disease resistance signaling proteins, EDS1 and PAD4. The EMBO Journal 20:5400-11
51. Flor HH. 1955. Host-parasite interaction in flax rust—its genetics and other implications. Phytopathology 45:680-5
52. Frost D, Way H, Howles P, Luck J, Manners J, et al. 2004. Tobacco transgenic for the flax rust resistance gene L expresses allele-specific activation of defense responses. Molecular Plant-Microbe Interactions 17:224-32
53. Giannakopoulou A, Steele JF, Segretin ME, Bozkurt TO, Zhou J, et al. 2015. Tomato I2 Immune Receptor Can Be Engineered to Confer Partial Resistance to the Oomycete Phytophthora infestans in Addition to the Fungus Fusarium oxysporum. Molecular Plant-Microbe Interactions 28:1316-29
54. González VM, Müller S, Baulcombe D, Puigdomènech P. 2015. Evolution of NBS-LRR gene copies among dicot plants and its regulation by members of the miR482/2118 superfamily of miRNAs. Molecular Plant 8:329-31
55. Gu L, Si W, Zhao L, Yang S, Zhang X. 2015. Dynamic evolution of NBS-LRR genes in bread wheat and its progenitors. Molecular Genetics Genomic 290:727-38
56. Guo YL, Fitz J, Schneeberger K, Ossowski S, Cao J, Weigel D. 2011. Genome-wide comparison of nucleotide-binding site-leucine-rich repeat-encoding genes in Arabidopsis. Plant Physiology 157:757-69
57. Gust AA, Pruitt R, Nurnberger T. 2017. Sensing danger: key to activating plant immunity. Trends in Plant Science 22:779-91
58. He XF, Fang YY, Feng L, Guo HS. 2008. Characterization of conserved and novel microRNAs and their targets, including a TuMV-induced TIR-NBS-LRR class R gene-derived novel miRNA in Brassica. FEBS Letters 582:2445-52
59. Horsefield S, Burdett H, Zhang X, Manik MK, Shi Y, et al. 2019. NAD(+) cleavage activity by animal and plant TIR domains in cell death pathways. Science 365:793-9
60. Huang S, van der Vossen EA, Kuang H, Vleeshouwers VG, Zhang N, et al. 2005. Comparative genomics enabled the isolation of the R3a late blight resistance gene in potato. Plant Journal 42:251-61
61. Huh SU, Cevik V, Ding P, Duxbury Z, Ma Y, et al. 2017. Protein-protein interactions in the RPS4/RRS1 immune receptor complex. PLoS Pathogens 13:e1006376
62. Jia Y, McAdams SA, Bryan GT, Hershey HP, Valent B. 2000. Direct interaction of resistance gene and avirulence gene products confers rice blast resistance. The EMBO Journal 19:4004-14
63. Jia Y, Yuan Y, Zhang Y, Yang S, Zhang X. 2015. Extreme expansion of NBS-encoding genes in Rosaceae. BMC Genetics 16:48
64. Jones JD, Vance RE, Dangl JL. 2016. Intracellular innate immune surveillance devices in plants and animals. Science 354:aaf6395
65. Jubic LM, Saile S, Furzer OJ, El Kasmi F, Dangl JL. 2019. Help wanted: helper NLRs and plant immune responses. Current Opinion in Plant Biology 50:82-94
66. Karasov TL, Kniskern JM, Gao L, DeYoung BJ, Ding J, et al. 2014. The long-term maintenance of a resistance polymorphism through diffuse interactions. Nature 512:436-40
67. Kim S, Park J, Yeom SI, Kim YM, Seo E, et al. 2017. New reference genome sequences of hot pepper reveal the massive evolution of plant disease-resistance genes by retroduplication. Genome Biology 18:210
68. Kim SH, Qi D, Ashfield T, Helm M, Innes RW. 2016. Using decoys to expand the recognition specificity of a plant disease resistance protein. Science 351:684-7
69. Krasileva KV, Dahlbeck D, Staskawicz BJ. 2010. Activation of an Arabidopsis resistance protein is specified by the in planta association of its leucine-rich repeat domain with the cognate oomycete effector. Plant Cell 22:2444-58
70. Kroj T, Chanclud E, Michel-Romiti C, Grand X, Morel JB. 2016. Integration of decoy domains derived from protein targets of pathogen effectors into plant immune receptors is widespread. New Phytologist 210:618-26
71. Lapin D, Kovacova V, Sun X, Dongus JA, Bhandari D, et al. 2019. A coevolved EDS1-SAG101-NRG1 module mediates cell death signaling by TIR-domain immune receptors. Plant Cell 31:2430-55
72. Leister D. 2004. Tandem and segmental gene duplication and recombination in the evolution of plant disease resistance genes. Trends in Genetics 20:116-22
73. Lewis JD, Lee AH, Hassan JA, Wan J, Hurley B, et al. 2013. The Arabidopsis ZED1 pseudokinase is required for ZAR1-mediated immunity induced by the Pseudomonas syringae type III effector HopZ1a. PNAS 110:18722-7
74. Li F, Pignatta D, Bendix C, Brunkard JO, Cohn MM, et al. 2012. MicroRNA regulation of plant innate immune receptors. PNAS 109:1790-5
75. Li L, Habring A, Wang K, Weigel D. 2019. Oligomerization of the NLR immune receptor RPP7 triggered by the atypical resistance protein RPW8/HR as a ligand. bioRxiv:682807
76. Li Y, Zhong Y, Huang K, Cheng Z. 2017. Genomewide analysis of NBS-encoding genes in kiwi fruit (Actinidia chinensis). Journal of Genetics 95:997-1001
77. Liang W, van Wersch S, Tong M, Li X. 2019. TIR-NB-LRR immune receptor SOC3 pairs with truncated TIR-NB protein CHS1 or TN2 to monitor the homeostasis of E3 ligase SAUL1. New Phytologist 221:2054-66
78. Liang X, Zhou JM. 2018. Receptor-like cytoplasmic kinases: central players in plant receptor kinase-mediated signaling. Annual Review of Plant Biology 69:267-99
79. Lin X, Zhang Y, Kuang H, Chen J. 2013. Frequent loss of lineages and deficient duplications accounted for low copy number of disease resistance genes in Cucurbitaceae. BMC Genomics 14:335
80. Liu C, Cui D, Zhao J, Liu N, Wang B, et al. 2019. Two Arabidopsis receptor-like cytoplasmic kinases SZE1 and SZE2 associate with the ZAR1-ZED1 complex and are required for effector-triggered immunity. Molecular Plant 12:967-83
81. Liu J, Cheng X, Liu D, Xu W, Wise R, Shen QH. 2014. The miR9863 family regulates distinct Mla alleles in barley to attenuate NLR receptor-triggered disease resistance and cell-death signaling. PLoS Genetics 10:e1004755
82. Lu X, Kracher B, Saur IM, Bauer S, Ellwood SR, et al. 2016. Allelic barley MLA immune receptors recognize sequence-unrelated avirulence effectors of the powdery mildew pathogen. PNAS 113:E6486-E95
83. Luo S, Zhang Y, Hu Q, Chen J, Li K, et al. 2012. Dynamic nucleotide-binding site and leucine-rich repeat-encoding genes in the grass family. Plant Physiology 159:197-210
84. Ma Y, Guo H, Hu L, Martinez PP, Moschou PN, et al. 2018. Distinct modes of derepression of an Arabidopsis immune receptor complex by two different bacterial effectors. PNAS 115:10218-27
85. Mackey D, Belkhadir Y, Alonso JM, Ecker JR, Dangl JL. 2003. Arabidopsis RIN4 is a target of the Type III virulence effector AvrRpt2 and modulates RPS2-mediated resistance. Cell 112:379-89
86. Mackey D, Holt BF, Wiig A, Dangl JL. 2002. RIN4 interacts with Pseudomonas syringae Type III effector molecules and is required for RPM1-mediated resistance in Arabidopsis. Cell 108:743-54
87. Maekawa T, Cheng W, Spiridon LN, Toller A, Lukasik E, et al. 2011. Coiled-coil domain-dependent homodimerization of intracellular barley immune receptors defines a minimal functional module for triggering cell death. Cell Host Microbe 9:187-99
88. Mago R, Zhang P, Vautrin S, Simkova H, Bansal U, et al. 2015. The wheat Sr50 gene reveals rich diversity at a cereal disease resistance locus. Nature Plants 1:15186
89. Maqbool A, Saitoh H, Franceschetti M, Stevenson CE, Uemura A, et al. 2015. Structural basis of pathogen recognition by an integrated HMA domain in a plant NLR immune receptor. Elife 4:e08709
90. Meyers BC, Kozik A, Griego A, Kuang H, Michelmore RW. 2003. Genome-wide analysis of NBS-LRR-encoding genes in Arabidopsis. Plant Cell 15:809-34
91. Michelmore RW, Meyers BC. 1998. Clusters of resistance genes in plants evolve by divergent selection and a birth-and-death process. Genome Research 8:1113-30
92. Mondragon-Palomino M, Gaut BS. 2005. Gene conversion and the evolution of three leucine-rich repeat gene families in Arabidopsis thaliana. Molecular Biology Evolution 22:2444-56
93. Nam KH, Li J. 2002. BRI1/BAK1, a receptor kinase pair mediating brassinosteroid signaling. Cell 110:203-12
94. Nandety RS, Caplan JL, Cavanaugh K, Perroud B, Wroblewski T, et al. 2013. The role of TIR-NBS and TIR-X proteins in plant basal defense responses. Plant Physiology 162:1459-72
95. Narusaka M, Kubo Y, Shiraishi T, Iwabuchi M, Narusaka Y. 2009. A dual resistance gene system prevents infection by three distinct pathogens. Plant Signaling Behavior 4:954-5
96. Nishimura MT, Anderson RG, Cherkis KA, Law TF, Liu QL, et al. 2017. TIR-only protein RBA1 recognizes a pathogen effector to regulate cell death in Arabidopsis. PNAS 114:E2053-E62
97. Nuthikattu S, McCue AD, Panda K, Fultz D, DeFraia C, et al. 2013. The initiation of epigenetic silencing of active transposable elements is triggered by RDR6 and 21-22 nucleotide small interfering RNAs. Plant Physiology 162:116-31
98. Ori N, Eshed Y, Paran I, Presting G, Aviv D, et al. 1997. The I2C family from the wilt disease resistance locus I2 belongs to the nucleotide binding, leucine-rich repeat superfamily of plant resistance genes. Plant Cell 9:521-32
99. Ouyang S, Park G, Atamian HS, Han CS, Stajich JE, et al. 2014. MicroRNAs suppress NB domain genes in tomato that confer resistance to Fusarium oxysporum. PLoS Pathogens 10:e1004464
100. Paoletti M, Clave C. 2007. The fungus-specific HET domain mediates programmed cell death in Podospora anserina. Eukaryotic Cell 6:2001-8
101. Petre B, Saunders DG, Sklenar J, Lorrain C, Krasileva KV, et al. 2016. Heterologous expression screens in Nicotiana benthamiana identify a candidate effector of the wheat yellow rust pathogen that associates with processing bodies. PLoS One 11:e0149035
102. Qi T, Seong K, Thomazella DPT, Kim JR, Pham J, et al. 2018. NRG1 functions downstream of EDS1 to regulate TIR-NLR-mediated plant immunity in Nicotiana benthamiana. PNAS 115:E10979-E87
103. Rodin SN, Riggs AD. 2003. Epigenetic silencing may aid evolution by gene duplication. Journal of Molecular Evolution 56:718-29
104. Sandstrom A, Mitchell PS, Goers L, Mu EW, Lesser CF, Vance RE. 2019. Functional degradation: a mechanism of NLRP1 inflammasome activation by diverse pathogen enzymes. Science 364:eaau1330
105. Sarris PF, Cevik V, Dagdas G, Jones JD, Krasileva KV. 2016. Comparative analysis of plant immune receptor architectures uncovers host proteins likely targeted by pathogens. BMC Biology 14:8
106. Sarris PF, Duxbury Z, Huh SU, Ma Y, Segonzac C, et al. 2015. A Plant immune receptor detects pathogen effectors that target WRKY transcription factors. Cell 161:1089-100
107. Saucet SB, Ma Y, Sarris PF, Furzer OJ, Sohn KH, Jones JD. 2015. Two linked pairs of Arabidopsis TNL resistance genes independently confer recognition of bacterial effector AvrRps4. Nature Communications 6:6338
108. Schultink A, Qi T, Bally J, Staskawicz B. 2019. Using forward genetics in Nicotiana benthamiana to uncover the immune signaling pathway mediating recognition of the Xanthomonas perforans effector XopJ4. New Phytologist 221:1001-9
109. Segretin ME, Pais M, Franceschetti M, Chaparro-Garcia A, Bos JI, et al. 2014. Single amino acid mutations in the potato immune receptor R3a expand response to Phytophthora effectors. Molecular Plant-Microbe Interactions 27:624-37
110. Seo E, Kim T, Park JH, Yeom SI, Kim S, et al. 2018. Genome-wide comparative analysis in Solanaceous species reveals evolution of microRNAs targeting defense genes in Capsicum spp. DNA Research 25:561-75
111. Serra H, Choi K, Zhao X, Blackwell AR, Kim J, Henderson IR. 2018. Interhomolog polymorphism shapes meiotic crossover within the Arabidopsis RAC1 and RPP13 disease resistance genes. PLoS Genetics 14:e1007843
112. Seto D, Koulena N, Lo T, Menna A, Guttman DS, Desveaux D. 2017. Expanded type III effector recognition by the ZAR1 NLR protein using ZED1-related kinases. Nature Plants 3:17027
113. Shao ZQ, Xue JY, Wu P, Zhang YM, Wu Y, et al. 2016. Large-scale analyses of angiosperm nucleotide-binding site-leucine-rich repeat genes reveal three anciently diverged classes with distinct evolutionary patterns. Plant Physiology 170:2095-109
114. Shen QH, Zhou F, Bieri S, Haizel T, Shirasu K, Schulze-Lefert P. 2003. Recognition specificity and RAR1/SGT1 dependence in barley Mla disease resistance genes to the powdery mildew fungus. Plant Cell 15:732-44
115. Shivaprasad PV, Chen HM, Patel K, Bond DM, Santos BA, Baulcombe DC. 2012. A microRNA superfamily regulates nucleotide binding site-leucine-rich repeats and other mRNAs. Plant Cell 24:859-74
116. Simons G, Groenendijk J, Wijbrandi J, Reijans M, Groenen J, et al. 1998. Disp of the Fusarium I2 gene cluster in tomato reveals six homologs and one active gene copy Plant Cell 10:1055-68
117. Slootweg E, Koropacka K, Roosien J, Dees R, Overmars H, et al. 2017. Sequence exchange between homologous NB-LRR genes converts virus resistance into nematode resistance, and vice versa. Plant Physiology 175:498-510
118. Song W, Wang B, Li X, Wei J, Chen L, et al. 2015. Identification of immune related LRR-containing genes in maize (Zea mays L.) by genome-wide sequence analysis. Internaional Journal of Genomics 2015:231358
119. Stam R, Scheikl D, Tellier A. 2016. Pooled Enrichment Sequencing Identifies Diversity and Evolutionary Pressures at NLR Resistance Genes within a Wild Tomato Population. Genome Biology and Evolution 8:1501-15
120. Stam R, Silva-Arias GA, Tellier A. 2019. Subsets ofNLR genes show differential signatures of adaptation during colonization of new habitats. New Phytologist 224:376-9
121. Stein JC, Yu Y, Copetti D, Zwickl DJ, Zhang L, et al. 2018. Genomes of 13 domesticated and wild rice relatives highlight genetic conservation, turnover and innovation across the genus Oryza. Nature Genetics 50:285-96
122. Swiderski MR, Birker D, Jones JD. 2009. The TIR domain of TIR-NB-LRR resistance proteins is a signaling domain involved in cell death induction. Molecular Plant-Microbe Interactions 22:157-65
123. Takken FL, Albrecht M, Tameling WI. 2006. Resistance proteins: molecular switches of plant defence. Current Opinion in Plant Biology 9:383-90
124. Takken FL, Goverse A. 2012. How to build a pathogen detector: structural basis of NB-LRR function. Current Opinion in Plant Biology 15:375-84
125. Tameling WI, Elzinga SD, Darmin PS, Vossen JH, Takken FL, et al. 2002. The tomato R gene products I-2 and MI-1 are functional ATP binding proteins with ATPase activity. Plant Cell 14:2929-39
126. Tenthorey JL, Haloupek N, López-Blanco JR, Grob P, Adamson E, et al. 2017. The structural basis of flagellin detection by NAIP5: A strategy to limit pathogen immune evasion Science 358:888-93
127. Urbach JM, Ausubel FM. 2017. The NBS-LRR architectures of plant R-proteins and metazoan NLRs evolved in independent events. PNAS 114:1063-8
128. Van de Weyer AL, Monteiro F, Furzer OJ, Nishimura MT, Cevik V, et al. 2019. A species-wide inventory of NLR genes and alleles in Arabidopsis thaliana. Cell 178:1260-72
129. van der Hoorn RA, Kamoun S. 2008. From guard to decoy: a new model for perception of plant pathogen effectors. Plant Cell 20:2009-17
130. Vleeshouwers VG, Raffaele S, Vossen JH, Champouret N, Oliva R, et al. 2011. Understanding and exploiting late blight resistance in the age of effectors. Annuai Review of Phytopathology 49:507-31
131. Wan L, Essuman K, Anderson RG, Sasaki Y, Monteiro F, et al. 2019. TIR domains of plant immune receptors are NAD(+)-cleaving enzymes that promote cell death. Science 365:799-803
132. Wang G, Roux B, Feng F, Guy E, Li L, et al. 2015. The decoy substrate of a pathogen effector and a pseudokinase specify pathogen-induced modified-self recognition and immunity in plants. Cell Host Microbe 18:285-95
133. Wang J, Hu M, Wang J, Qi J, Han Z, et al. 2019. Reconstitution and structure of a plant NLR resistosome conferring immunity. Science 364:eaav5870
134. Wang J, Wang J, Hu M, Wu S, Qi J, et al. 2019. Ligand-triggered allosteric ADP release primes a plant NLR complex. Science 364:eaav5868
135. Wang W, Devoto A, Turner JG, Xiao S. 2007. Expression of the membrane-associated resistance protein RPW8 enhances basal defense against biotrophic pathogens. Molecular Plant-Microbe Interactions 20:966-76
136. Wessling R, Epple P, Altmann S, He Y, Yang L, et al. 2014. Convergent targeting of a common host protein-network by pathogen effectors from three kingdoms of life. Cell Host Microbe 16:364-75
137. Williams SJ, Sornaraj P, deCourcy-Ireland E, Menz R, Kobe B, et al. 2011. An autoactive mutant of the M flax rust resistance protein has a preference for binding ATP, whereas wild-type M protein binds ADP. molecular Plant-Microbe Interactions 24:897-906
138. Williams SJ, Yin L, Foley G, Casey LW, Outram MA, et al. 2016. Structure and function of the TIR domain from the grape NLR protein RPV1. Frontiers in Plant Science 7:1850
139. Wroblewski T, Spiridon L, Martin EC, Petrescu AJ, Cavanaugh K, et al. 2018. Genome-wide functional analyses of plant coiled-coil NLR-type pathogen receptors reveal essential roles of their N-terminal domain in oligomerization, networking, and immunity. PLoS Biology 16:e2005821
140. Wu CH, Abd-El-Haliem A, Bozkurt TO, Belhaj K, Terauchi R, et al. 2017. NLR network mediates immunity to diverse plant pathogens. PNAS 114:8113-8
141. Wu CH, Belhaj K, Bozkurt TO, Birk MS, Kamoun S. 2016. Helper NLR proteins NRC2a/b and NRC3 but not NRC1 are required for Pto-mediated cell death and resistance in Nicotiana benthamiana New Phytologist 209:1344-52
142. Wu CH, Kamoun S. 2019. Tomato Prf requires NLR helpers NRC2 and NRC3 to confer resistance against the bacterial speck pathogen Pseudomonas syringae pv. tomato. bioRxiv:595744
143. Wu Z, Li M, Dong OX, Xia S, Liang W, et al. 2019. Differential regulation of TNL-mediated immune signaling by redundant helper CNLs. New Phytologist 222:938-53
144. Xia R, Xu J, Arikit S, Meyers BC. 2015. Extensive families of miRNAs and PHAS loci in Norway spruce demonstrate the origins of complex phasiRNA networks in seed plants. Molecular Biology Evolution 32:2905–18
145. Xiao S, Ellwood S, Calis O, Patrick E, Li T, et al. 2001. Broad-spectrum mildew resistance in Arabidopsis thaliana mediated by RPW8. Science 291:118-20
146. Yoshida K, Schuenemann VJ, Cano LM, Pais M, Mishra B, et al. 2013. The rise and fall of the Phytophthora infestans lineage that triggered the Irish potato famine. Elife 2:e00731
147. Zhai J, Jeong DH, De Paoli E, Park S, Rosen BD, et al. 2011. MicroRNAs as master regulators of the plant NB-LRR defense gene family via the production of phased, trans-acting siRNAs. Genes Development 25:2540-53
148. Zhang X, Bernoux M, Bentham AR, Newman TE, Ve T, et al. 2017. Multiple functional self-association interfaces in plant TIR domains. PNAS 114:E2046-E52
149. Zhang Y, Xia R, Kuang H, Meyers BC. 2016. The diversification of plant NBS-LRR defense genes directs the evolution of microRNAs that target them. Molecular Biology Evolution 33:2692-705
150. Zhang Z, Wu Y, Gao M, Zhang J, Kong Q, et al. 2012. Disruption of PAMP-induced MAP kinase cascade by a Pseudomonas syringae effector activates plant immunity mediated by the NB-LRR protein SUMM2. Cell Host Microbe 11:253-63
151. Zhou M, Li Y, Hu Q, Bai XC, Huang W, et al. 2015. Atomic structure of the apoptosome: mechanism of cytochrome c- and dATP-mediated activation of Apaf-1. Genes Development 29:2349-61
来源:PlantBiotech 植物生物学
原文链接:http://mp.weixin.qq.com/s?__biz=MzI5NTk2MTcyOA==&mid=2247493323&idx=1&sn=58e8c8d13c97ac1187e09236fd320e54
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
先祖性墨西哥类蜀黍等位基因可导致更高产的密集种植玉米

Nature Plants :刘磊博士等利用基因编辑技术创制玉米高产等位基因
研究揭示新等位基因动态调控棉花纤维伸长分子机理
BMC Bioinformatics基于癌细胞等位基因频率改变的生殖系单倍体分型

南京农大洪德林团队鉴定到一个水稻灌浆率相关的有利等位基因

Nuc Acid Res:单细胞水平揭示等位基因对雌激素的反应性

中科院遗传发育所完成大规模中国水稻品系重要农艺性状遗传结构及有利等位基因应用分析

变异的风险等位基因左右精神疾病遗传机制

新的等位基因让水稻更好吃

研究发现参与导致小麦赤霉病的新分子