来源:植物科学最前沿
今天,给大家推送发表在“植物学报”的一篇方法论文,在本文中, 详细地介绍GST标签融合蛋白的Pull-down 和烟草瞬时表达系统中Co-IP两种技术的实验流程, 为验证植物中蛋白的相互作用提供参考。该论文作者为徐重益研究员,正在做相关实验的同学可以下载全文学习一下~摘要: 蛋白相互作用在细胞的生命活动中起着关键作用,在不同时空层面上参与数个细胞过程。因此,研究蛋白相互作用对于理解分子调控网络至关重要。通常情况下,利用酵母双杂交系统筛选的植物蛋白相互作用必须利用体外和体内系统进行验证。Pull-down和Co-IP就是验证植物蛋白相互作用的常用技术。Pull-down被广泛用于体外验证蛋白间的直接相互作用,而在植物活体内,利用本氏烟草叶片瞬时表达蛋白继而采用Co-IP鉴定是目前验证蛋白相互作用最简单最快速的方法之一。本文就GST Pull-down和烟草瞬时表达系统中Co-IP技术的原理和实验方案进行详细描述,以期为验证植物蛋白相互作用提供方法上的参考。
酵母双杂交系统对于研究植物蛋白间相互作用是非常有用的(Stasi et al., 2015)。具体而言, 它是一项筛选和发现新互作蛋白的首选技术。然而, 由于酵母本身是个异源系统, 存在体内无法正常重建蛋白互作原始环境的缺陷。例如, 酵母双杂交技术迫使两个蛋白同时定位于细胞核中, 但植物中原始蛋白存在于不同的细胞结构中, 于是会导致一些假阳性结果因此, 酵母双杂交系统获得的候选蛋白必须通过一些其它方法, 尤其需要在相似的生物体中进行验证。
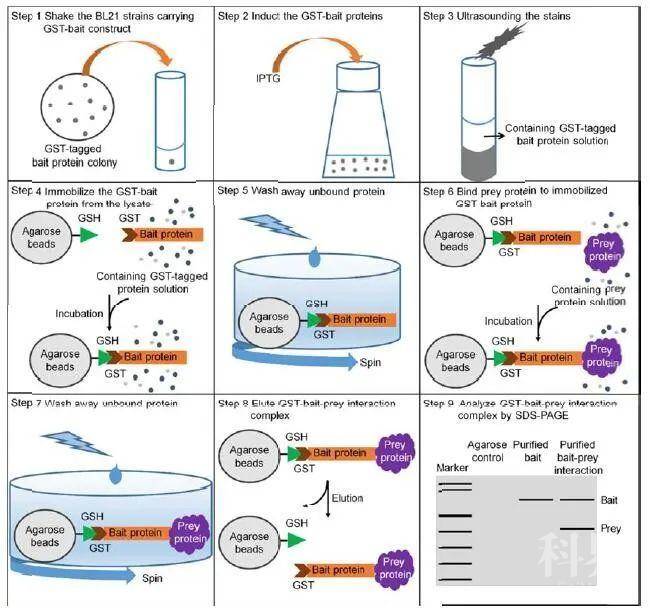
Pull-down 技术, 也叫蛋白质体外结合实验, 是一种行之有效的验证酵母双杂交系统的体外试验技术(Wissmueller et al., 2011)。早在1988 年, Smith 和Johnson (1988)利用谷胱甘肽-S-转移酶(GST)融合标签从细菌中一步纯化出GST 融合蛋白。从此GST 融合蛋白在蛋白质相互作用研究领域里得到了极大的推广。
利用GST 融合蛋白进行Pull-down 的基本原理是将靶蛋白-GST 融合蛋白亲和固化在谷胱甘肽标记的珠子上, 作为与目的蛋白亲和的支撑物, 充当“诱饵蛋白”, 目的蛋白溶液与之孵育, 可从中捕获与之相互作用的“捕获蛋白”(目的蛋白), 洗脱结合物后通过SDS-PAGE (Sodium Dodecyl Sulfate-Polyacrylamide Gel Electrophoresis)电泳分析, 从而证实两种蛋白间的相互作用或筛选相应的目的蛋白,诱饵蛋白和“捕获蛋白”均可通过细胞裂解物、纯化的蛋白、表达系统以及体外转录翻译系统等方法获得。
在Pull-down 实验中, 设计恰当的对照实验非常重要, 该对照可以是表达有GST (非诱饵融合蛋白)的转化细胞裂解物, 也可以是将GST 加到非转化细胞的裂解物中。GST 融合蛋白Pull-down 的方法可以用来鉴定能与已知融合蛋白相互作用的未知蛋白质, 也可以鉴定2 个已知蛋白质之间是否存在相互作用。此方法简单易行, 操作方便。但Pull-down 的结果并不能真实的反应蛋白质之间的相互作用, 因为在活体内它们不一定在亚细胞空间上能碰到, 所以并不意味着在生理条件下一定结合。
植物中许多蛋白之间的相互作用是通过酵母双杂交技术筛选发现的, 在大多数情况下,由于获得表达两种蛋白的稳定转化植株是一个费时又费力的过程, 要将这些蛋白共表达于自身的植物物种中并进行互作验证并不容易。在这种情况下, 我们可以采用一种瞬时表达系统来快速表达目的蛋白并检测蛋白互作。
目前, 最常用且较稳定的植物瞬时表达系统是用含有表达假定相互作用蛋白质粒的农杆菌(Agrobacterium tumefaciens)菌株混合物注射本氏烟草(Nicotiana benthamiana)的叶片(Hirsch et al., 2009; Fernandez-Bautista et al., 2017;Munoz et al., 2017)。
一旦植物蛋白被瞬时表达, 检测互作的方法一般采用免疫共沉淀(Co-immunoprecipitation, Co-IP) (Takahashi, 2015)。Co-IP是以抗体和抗原之间的专一性作用为基础来研究蛋白质相互作用的经典方法, 也是确定两种蛋白在完整细胞内生理性相互作用的有效方法。其原理是: 当细胞在非变性条件下被裂解时, 完整细胞内存在的蛋白相互作用得以保持, 当蛋白粗提物与能够识别目的蛋白的琼脂糖凝胶交联的标签抗体珠子(agarose-conjugated anti-TAG beads)一起孵育时, 与目的蛋白在体内结合的互作蛋白也被沉淀下来。这里通常采用目的蛋白融合一个商业化的标签以特异识别标签抗体珠子来实现,使用较多的商业化标签主要有HA (hemagglutinin)、Flag或c-Myc肽段。
将粗提物与珠子一起温育后, 用相同的提取缓冲液洗涤数次, 以消除所有未与珠子结合的蛋白质, 同时保留特定的互作蛋白。接着从珠子中洗脱蛋白质复合物, 最直接的方法是用电泳上样缓冲液煮沸珠子。最后, 用SDS-PAGE检测等份的粗提物(以分析所产生的蛋白质的表达水平)和洗脱产物(以检测目的蛋白之一的免疫沉淀和互作蛋白的免疫共沉淀), 使用特异性标签抗体进行蛋白质印迹。
这种方法常用于测定两种目的蛋白是否在体内结合; 也可用于确定一种特定蛋白新的互作蛋白。该方法的优点在于相互作用的蛋白都是经翻译后修饰的, 处于天然状态; 蛋白的相互作用是在自然状态下进行的, 可以避免人为的影响; 可以分离得到天然状态下相互作用的蛋白复合物。缺点: 可能检测不到低亲和力和蛋白与蛋白之间的瞬时相互作用; 两种蛋白可能不是直接结合, 而需要有起桥梁作用的第3个蛋白存在; 必须在实验前预测目的蛋白是什么, 以选择最后检测的抗体。
Pull-down 与Co-IP 的原理是类似的, 都是利用亲和配体来捕获互作蛋白, 所不同的是,Pull-down 是利用一个纯化且含有标签的蛋白作为诱饵来结合互作蛋白, 一般用来证明蛋白之间直接的相互作用; 而Co-IP 则采用固定的抗体捕获互作蛋白, 用来证明2 个蛋白之间的相互作用, 但不排除通过第3 个蛋白介导的间接相互作用。在本文中, 我们将详细地介绍GST标签融合蛋白的Pull-down 和烟草瞬时表达系统中Co-IP两种技术的实验流程, 以期为验证植物中蛋白的相互作用提供参考。
原文链接:http://www.chinbullbotany.com/CN/10.11983/CBB19143
来源:frontiersin 植物科学最前沿
原文链接:https://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247494257&idx=1&sn=93baa3cf90010753b183d96af8c30f73&chksm=e8bd986fdfca11791514be9a6aed4b0beceb2c448c57d6a03bcbf544cbc4dd19fbe7ed0a2288#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn