科技工作者之家
科界APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-03-13
来源:BioArt
原标题:Cell 付向东实验室揭示精卵结合后精子DNA起始解压缩及染色质重构的重要机制
当精子与卵细胞结合后,新生命迅速开启一系列染色质重编程程序,从而实现自身基因组激活(受精卵基因组激活,ZGA)。近年来,随着基因组学技术(Genomic Profiling)的广泛应用,我们对这一胚胎早期发育过程各个阶段的生物学事件有了长足的认识和理解,但受精卵如何在第一时间启动染色质重编程仍是发育生物学领域内长久以来的未解之迷。
追溯到精子发生后期,单倍体精子细胞的全基因组将逐步发生剧烈的染色质重构,与DNA结合的组蛋白几乎全部被一种富含精氨酸的碱性核蛋白——鱼精蛋白(Protamine)所替换(“组蛋白 - 鱼精蛋白置换”),从而造成基因转录活动逐渐停止、基因组高度折叠,最终使精子细胞核呈极度压缩状态,这一过程旨在确保精子将DNA信息安全、精确、无损地传递给后代。
而在受精之后,合子染色质重构的首要任务便是对精子携带入卵细胞的父源基因组进行快速、高效的解压缩,即用母源组蛋白替换包裹精子DNA的鱼精蛋白(“鱼精蛋白- 组蛋白置换”)。过去已有的研究表明,一类在卵细胞中高度表达的酸性蛋白因子nucleoplasmin(NPM2)在这一过程中参与将鱼精蛋白由父源染色质中移除【1-6】;同时,另一种母源组蛋白伴侣因子HIRA负责将组蛋白H3.3装配到父源DNA上【7-9】。但是,受精卵如何快速高效地启动父源染色质的“鱼精蛋白- 组蛋白置换”,在领域内一直鲜为人知。
鱼精蛋白是一类由组蛋白进化而来的富含精氨酸的小核蛋白,相对于常规组蛋白,鱼精蛋白带有更强的正电荷,因此其与DNA具有更强的结合能力。有趣的是,哺乳动物鱼精蛋白具有多个潜在的磷酸化修饰位点,且早年间的生化研究表明,多种激酶均能在体外条件下磷酸化修饰鱼精蛋白【10-12】。但是,鱼精蛋白的磷酸化修饰具有何种生物学功能?这种修饰在体内是否可能成为调控鱼精蛋白与DNA亲和力的一种主要方式?如果是,在体内究竟由何种激酶来负责催化鱼精蛋白?如果仔细对鱼精蛋白的蛋白质序列进行观察分析,不难发现哺乳动物鱼精蛋白具有若干个潜在且保守的丝氨酸(Ser, S)磷酸化位点,且每一个丝氨酸均与相邻的精氨酸(Arg, R)构成 “Ser/Arg二肽”。有趣的是,这种特异的二肽也普遍存在于负责RNA剪接的SR蛋白中,且这类二肽序列特征能够被SR蛋白特异性剪接激酶SRPK所识别并进行磷酸化修饰。
美国加州大学圣地亚哥分校细胞与分子医学系付向东教授实验室于上世纪九十年代发现了剪接因子SR蛋白特异性的激酶家族(SRPK)【13-14】,并长期致力于SRPK激酶在pre-mRNA剪接过程中的功能机制研究,其一系列开创性工作均已成为当前SRPK激酶研究的关键文献资料。在本研究的前期工作中,付向东实验室苟兰涛博士发现雌性小鼠MII 卵细胞中富含大量的SRPK1蛋白,且体外磷酸化实验证明SRPK1激酶可高效催化化学合成的小鼠鱼精蛋白1 (Protamine 1, P1),提示卵细胞内广泛分布的母源SRPK1激酶可能通过催化进入卵细胞内的精子染色质中的鱼精蛋白来调控启动受精卵早期染色质重构。
2020年3月12日,付向东实验室在Cell上发表文章 Initiation of Parental Genome Reprogramming in Fertilized Oocyte by Splicing Kinase SRPK1-Catalyzed Protamine Phosphorylation ,首次报道了SR蛋白特异性剪接激酶SRPK1在小鼠受精卵早期发育过程中具有重要功能:即母源SRPK1激酶负责催化进入卵细胞的精子DNA快速起始解压缩,协助启动受精卵早期染色质重构,从而保障雄原核与雌原核逐步发育形成并随后融合。
为了探究卵细胞内的母源SRPK1究竟是否在受精卵及早期胚胎发育中发挥重要生物学功能,该研究作者首先利用SRPK1特异性抑制剂【15】处理小鼠超排的MII卵细胞,发现该抑制剂处理不会造成卵细胞体外受精率的显著改变,但可导致部分受精卵发育阻滞停留在1细胞期。随后,作者进一步通过 SRPK1敲除小鼠模型证实该激酶的确在受精卵发育过程中扮演重要角色,且丧失SRPK1激酶很可能直接造成受精卵雄原核发育异常。
为了直接证明SRPK1激酶的催化活性为受精卵发育所必需,作者又利用小鼠卵细胞显微注射及体外培养技术【7, 16】,通过注射特异性siRNA将母源SRPK1敲低。这项实验进一步发现该激酶在受精卵内的缺失将造成精子染色质解压缩失败,雄原核发育停滞。有趣的是,如果在卵细胞显微操作时同时注入人源SRPK1 mRNA (拮抗siRNA作用),则受精卵及雄原核发育缺陷可被有效拯救恢复;而丧失激酶催化活性的人源SRPK1 mRNA (KD) 则没有拯救效果。因此,作者先后利用化学抑制剂处理、遗传学敲除、以及显微注射敲低与mRNA拯救这三种不同的实验策略,充分证明了母源SRPK1激酶的催化活性在受精卵父源染色质解压缩及重构过程中发挥重要功能。
随后,为了继续研究母源SRPK1激酶的作用途径和底物,作者制备并纯化了一系列鱼精蛋白磷酸化抗体,并进而证实了SRPK1激酶高效、迅速地催化受精后进入卵细胞的精子染色质,使其中的鱼精蛋白被磷酸化修饰。且体外、体内实验证据均表明鱼精蛋白1(P1)具有两个磷酸化修饰位点,分别位于其蛋白N端(Ser9)与C端(Ser 43)。接下来,为了进一步探究母源SRPK1是否通过催化鱼精蛋白磷酸化修饰而直接影响受精卵父源染色质解压缩,作者利用CRISPR/Cas9技术构建了鱼精蛋白1的knock-in小鼠,分别对其磷酸化位点Ser9与Ser43进行突变,产生三株特异的knock-in小鼠模型P1S9A、P1S43A、及 P1S9S43A。值得注意的是,单一磷酸化位点突变的精子(P1S9A、P1S43A)在进入卵细胞后仍可被正常解压缩并发生染色质重构,而不影响雄原核生成和受精卵发育;但磷酸化位点双突变的精子(P1S9S43A)虽然仍具有精子活性和功能,但其进入卵细胞后不能被有效解压缩,导致随后雄原核发育停滞。
那么,SRPK1介导的鱼精蛋白磷酸化修饰是如何启动父源染色质解压缩重构的呢?为了探究这一机制,作者利用体外实验系统研究并发现,化学合成的鱼精蛋白能够与双链DNA分子在体外迅速地发生相变分离(Phase transition),形成胶状颗粒(gel-like particle),并逐渐形成致密的交联网络。如果把这一过程简单地理解为精子发生过程中鱼精蛋白对基因组DNA的压缩排列,那么鱼精蛋白的磷酸化修饰会对其造成何种改变和影响呢?体外实验表明,SRPK1处理可“融化”致密排列的交联网络,逆转鱼精蛋白与DNA形成的高维结构。
进一步,作者用体外纯化的SRPK1激酶处理小鼠精子, 发现经过处理的小鼠精子能够更加高效地被NPM2作用并快速丧失鱼精蛋白。同时,作者还发现,鱼精蛋白磷酸化能够负责招募组蛋白伴侣HIRA富集于受精卵父源染色质并介导H3.3的装配。对上述实验现象的机制研究发现,鱼精蛋白磷酸化修饰后其与NPM2及HIRA的亲合力大大增强。因此,作者认为当精子染色质中的鱼精蛋白被磷酸化修饰后,其一方面协助打开染色质结构,同时促进招募NPM2与HIRA蛋白因子,协同负责染色质中鱼精蛋白的剔除与组蛋白H3.3的装配,最终实现高效“鱼精蛋白- 组蛋白置换”。
最后,作者进一步使用染色质开放性检测技术(ATAC-seq)调查受精后父源及母源染色质重构的进程。他们分别采用C57BL/6品系小鼠的精子与BALB/cJ品系小鼠的卵细胞进行体外受精,对处于早期发育阶段(PN2-PN3)的受精卵基因组中的开放区域进行检测记录,随后利用两种不同品系小鼠基因组中的SNP差异【17】进行后期数据拆分,即分别获得父源及母源染色质重构信息。与之前发现一致,与P1S9A及P1S43A单突变精子形成的受精卵,其早期父源、母源染色质开放区域均与野生型无异;而P1S9S43A双突变父源染色质基本完全继承保留了精子中的染色质开放状态。令人意外的是, P1S9S43A双突变父源染色质重构的停滞还造成了其母源染色质开放状态的异常,其完全保留了MII卵细胞的染色质开放区域,并未观察到其进行了明显的染色质重构。这一结果不但从全基因组角度再次证明SRPK1介导的鱼精蛋白磷酸化对受精卵染色质重构起到重要起始调控作用,还进一步表明受精卵中父源及母源染色质的发育重构是相互协调和影响的。
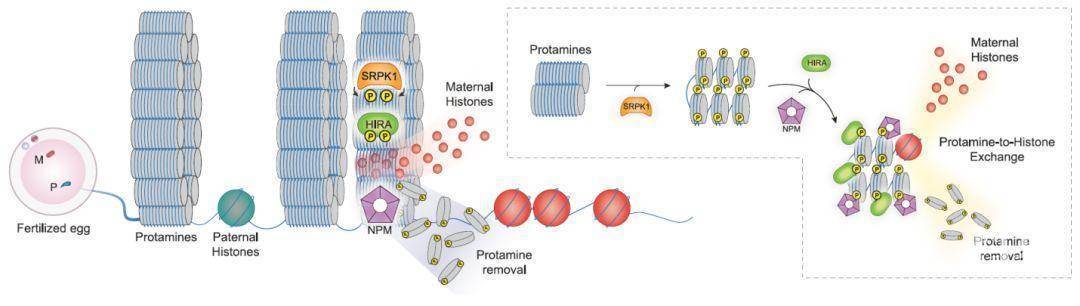
图1. 受精卵父源染色质解压缩及重构机制示意图
当前这项研究工作发现受精后母源SRPK1催化父源基因组鱼精蛋白磷酸化并启动染色质重构,不仅为解析早期受精卵染色质发育事件提供了新线索,还将有助于后期精子细胞染色质重构的深入研究,并为相关人类不育症的诊断治疗提供理论参考依据和靶点。
该项研究工作由美国加州大学圣地亚哥分校细胞与分子医学系付向东实验室完成,加州大学圣地亚哥分校苟兰涛博士、Do-Hwan Lim博士、马武彬博士为该研究论文的共同第一作者。此项研究同时得到了中国科学院分子细胞科学卓越创新中心(中科院生化细胞所)刘默芳研究员、加州大学圣地亚哥分校Joseph Adams教授、Pamela Mellon教授、Michael Rosenfeld教授、中国科学院分子细胞科学卓越创新中心(中科院生化细胞所)李党生研究员、中国科学院生物物理研究所许瑞明研究员等的大力协助。
参考文献
1. Inoue, A., Ogushi, S., Saitou, M., Suzuki, M.G., and Aoki, F. (2011). Involvement of Mouse Nucleoplasmin 2 in the Decondensation of Sperm Chromatin after Fertilization. Biol Reprod 85, 70-77.
2. Philpott, A., and Leno, G.H. (1992). Nucleoplasmin Remodels Sperm Chromatin in Xenopus Egg Extracts. Cell 69, 759-767.
3. Philpott, A., Leno, G.H., and Laskey, R.A. (1991). Sperm Decondensation in Xenopus Egg Cytoplasm Is Mediated by Nucleoplasmin. Cell 65, 569-578.
4. Shintomi, K., Takahashi, T.S., and Hirano, T. (2015). Reconstitution of mitotic chromatids with a minimum set of purified factors. Nat Cell Biol 17, 1014-U1336.
5. Shintomi, K., Inoue, F., Watanabe, H., Ohsumi, K., Ohsugi, M., and Hirano, T. (2017). Mitotic chromosome assembly despite nucleosome depletion in Xenopus egg extracts. Science 356, 1284-1287.
6. Okuwaki, M., Sumi, A., Hisaoka, M., Saotome-Nakamura, A., Akashi, S., Nishimura, Y., and Nagata, K. (2012). Function of homo- and hetero-oligomers of human nucleoplasmin/nucleophosmin family proteins NPM1, NPM2 and NPM3 during sperm chromatin remodeling. Nucleic Acids Res 40, 4861-4878.
7. Inoue, A., and Zhang, Y. (2014). Nucleosome assembly is required for nuclear pore complex assembly in mouse zygotes. Nat Struct Mol Biol 21, 609-616.
8. Lin, C.J., Koh, F.M., Wong, P., Conti, M., and Ramalho-Santos, M. (2014). Hira-Mediated H3.3 Incorporation Is Required for DNA Replication and Ribosomal RNA Transcription in the Mouse Zygote. Dev Cell 30, 268-279.
9. Loppin, B., Bonnefoy, E., Anselme, C., Laurencon, A., Karr, T.L., and Couble, P. (2005). The histone H3.3 chaperone HIRA is essential for chromatin assembly in the male pronucleus. Nature 437, 1386-1390.
10. Papoutsopoulou, S., Nikolakaki, E., Chalepakis, G., Kruft, V., Chevaillier, P., and Giannakouros, T. (1999). SR protein-specific kinase 1 is highly expressed in testis and phosphorylates protamine 1. Nucleic Acids Res 27, 2972-2980.
11. Pirhonen, A., Linnalakankkunen, A., and Maenpaa, P.H. (1994). P2 Protamines Are Phosphorylated in-Vitro by Protein-Kinase-C, Whereas P1 Protamines Prefer Camp-Dependent Protein-Kinase - a Comparative-Study of 5 Mammalian-Species. Eur J Biochem 223, 165-169.
12. Wu, J.Y., Ribar, T.J., Cummings, D.E., Burton, K.A., McKnight, G.S., and Means, A.R. (2000). Spermiogenesis and exchange of basic nuclear proteins are impaired in male germ cells lacking Camk4. Nat Genet 25, 448-452.
13. Gui, J.F., Lane, W.S., and Fu, X.D. (1994a). A Serine Kinase Regulates Intracellular-Localization of Splicing Factors in the Cell Cycle. Nature 369, 678-682.
14. Gui, J.F., Tronchere, H., Chandler, S.D., and Fu, X.D. (1994b). Purification and Characterization of a Kinase Specific for the Serine-Rich and Arginine-Rich Pre-Messenger-Rna Splicing Factors. Proc Natl Acad Sci USA 91, 10824-10828.
15. Hatcher, J.M., Wu, G., Zeng, C., Zhu, J., Meng, F., Patel, S., Wang, W., Ficarro, S.B., Leggett, A.L., Powell, C.E., et al. (2018). SRPKIN-1: A Covalent SRPK1/2 Inhibitor that Potently Converts VEGF from Pro-angiogenic to Anti-angiogenic Isoform. Cell Chem Biol 25, 460-470 e466.
16. Inoue, A., Sunaga, K., Aoki, F., and Zhang, Y. (2014). siRNA-Mediated Depletion of Stable Proteins in Mouse Oocytes. Protocol Exchange (July 21).
17. Link, V.M., Duttke, S.H., Chun, H.B., Holtman, I.R., Westin, E., Hoeksema, M.A., Abe, Y., Skola, D., Romanoski, C.E., Tao, J., et al. (2018). Analysis of Genetically Diverse Macrophages Reveals Local and Domain-wide Mechanisms that Control Transcription Factor Binding and Function. Cell 173, 1796-1809 e1717.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652482106&idx=1&sn=91af8c3485d2847f8ad1fc322166314c&chksm=84e2398eb395b098984f486b74fa9d4e3960c51e86ed19bdf0cfe192368c66f6199a097c4293&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
科学家发现“强势”染色体以“欺骗”手段进入卵细胞

中国科学院遗传发育所在植物磷酸化蛋白质组学技术研发方面获进展
精密测量院在磷酸化调控蛋白质结合机制研究中取得进展

北京大学陈鹏课题组和邹鹏课题组合作开发亚细胞磷酸化蛋白质组学新技术SubMAPP
染色质磷酸化蛋白质组学揭示PAMP触发免疫中核定位蛋白AHL13的功能!

克隆水稻来啦!

磷酸化调控蛋白质结合机制研究取得进展
人造卵巢支架让未成熟卵细胞正常发育
Dev Cell :刘延盛组发现磷酸化修饰对蛋白质表达稳定性的影响

周政课题组在组蛋白变体的染色质组装研究中取得新进展