科技工作者之家
科界APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-03-24
来源:BioArt
原标题:NSMB: 张锐组揭示ADAR蛋白全转录组RNA底物和决定ADAR结合和RNA编辑效率的特征
细胞内存在着多种功能重要的双链RNA结合蛋白,其中ADAR蛋白家族能结合到特定RNA的双链区域,并催化A碱基发生脱氨基反应生成I碱基,导致A-to-I RNA编辑【1】。A-to-I RNA编辑是动物中最普遍的RNA编辑类型,广泛的参与转录水平和转录后水平的多种基因调控机制,比如mRNA剪切、mRNA稳定性、环状RNA形成等等【2】。在哺乳动物细胞中,ADAR蛋白家族含有三个成员:ADAR1、ADAR2及ADAR3,它们在多器官发育、大脑功能中发挥重要的功能【1-2】。最近的研究表明ADAR1介导的非编码区域RNA编辑可用于标记“自我”和“非我”RNA,在先天免疫系统中起了重要的负调控作用(专家评述Nature+Nat Med丨RNA编辑酶ADAR1或可成为癌症免疫治疗新靶点)【3】。但是具体哪些ADAR底物是最关键的可以调控免疫反应的底物还不知道。由于RNA编辑的负调控效应,抑制ADAR1的编辑活性可以极大的促进肿瘤免疫疗法的效果,因此ADAR1可以作为肿瘤免疫疗法的重要靶标【4】。
此外,目前针对特定位点突变导致的疾病主要从基因组水平(比如CRISPR/Cas9单碱基编辑系统)修复。CRISPR/Cas9系统直接从基因组水平修复突变位点,但这一系统在DNA和RNA水平都有脱靶效应,并且Cas9蛋白本身会刺激细胞免疫反应,这些都限制了其临床应用。相比于DNA编辑,定点RNA编辑具有可控和可逆性,不影响基因组稳定性,并且可以只导入RNA而非蛋白,不会诱导免疫反应,在临床上具有一系列优势【5】。目前已有多个研究利用特定的guide RNA招募ADAR蛋白进行定点RNA编辑,但是这一领域最大的问题之一是RNA编辑效率低。揭示内源ADAR如何有效的识别和编辑底物可以用于指导和优化RNA靶向编辑系统。
尽管刻画ADAR蛋白的内源RNA底物在多个层面都具有重要的意义,但由于技术的局限性,目前研究人员对ADAR内源底物的特性及A-to-I RNA编辑的强度如何通过双链RNA的结构和碱基组成受到精细的调控所知甚少。
2020年3月23日,中山大学张锐课题组在Nature Structural & Molecular Biology杂志发表题为 irCLASH reveals RNA substrates recognized by human ADARs 的研究论文。该研究开发了一个高效的捕获RNA结合蛋白双链RNA底物的建库测序技术(irCLASH)以及后续的一整套生物信息学分析方法;首次在转录组水平上系统地绘制了人类ADAR蛋白的内源双链RNA底物图谱;揭示了决定ADAR结合效率和编辑效率的底物特征和ADAR结合长双链RNA的体内模型。

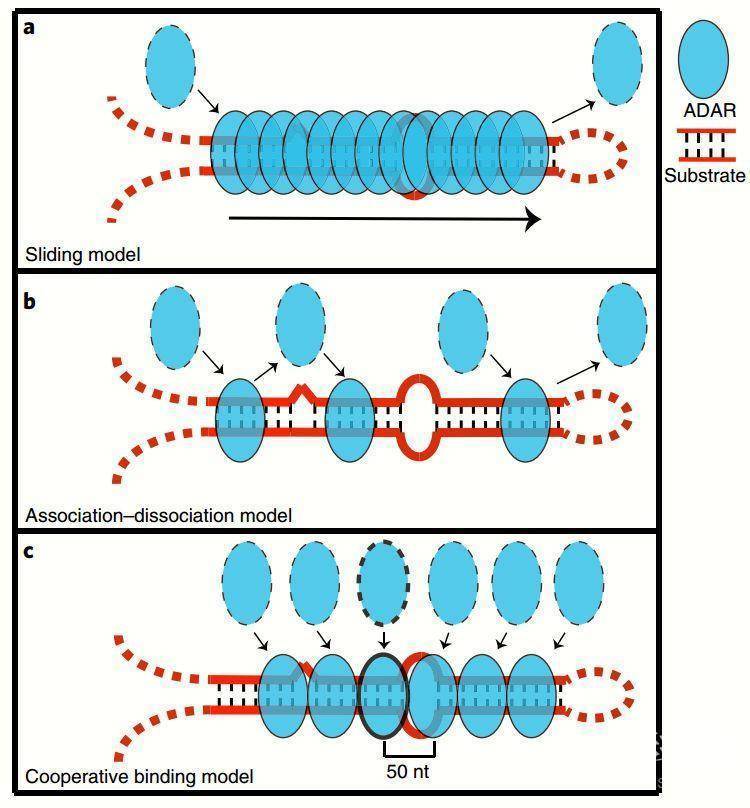
irCLASH技术可以在2天内完成整个建库流程,而且有较高的双链RNA底物捕获效率。作者利用irCLASH技术系统的构建和分析了人类ADAR1、ADAR2及ADAR3的双链RNA底物图谱,发现与之前根据计算推导的研究设想不同,ADAR具有大量的、长距离的双链RNA底物。作者还发现不完全互补配对的底物跟ADAR蛋白也有很好的亲和度,特别是ADAR2家族成员。一般来说,一个底物双臂之间的距离越长,其底物的双链结合区域就越长,但是双臂的距离长度跟底物的配对程度没有太大的相关性。通过对全转录组水平ADAR底物的一系列生物信息学分析及实验验证,作者进一步揭示了决定ADAR结合效率和编辑效率的底物特征,包括一些已知的通过体外底物实验推导出来的特征和一些之前所不了解的新特征。
另外,作者意外地发现ADAR蛋白能够串联结合于双链RNA底物上,并且相邻两个蛋白分子间大约间隔50bp;通过这种串联结合可以提高其RNA底物的编辑效率。基于上述发现,作者提出了一个ADAR结合长双链RNA的协同结合模型(见图),这一模型为长双链RNA编辑效率较高这一现象提供了一个可能的分子水平的解释。
最后,因为ADAR1和ADAR2蛋白主要结合前体mRNA,利用ADAR结合的全转录组水平的双链RNA信息,作者刻画出前体mRNA的结构。通过与之前发表的成熟mRNA结构的比较,揭示了前体mRNA和成熟mRNA截然不同的高级结构特性。
总之,作者通过开发irCLASH测序技术及后续的整套生物信息学分析方法为研究双链RNA结合蛋白的内源底物提供了新的工具。利用这一工具,作者捕获和刻画了ADAR蛋白家族每个成员的内源双链RNA底物,揭示了决定RNA编辑位置和强度的特征,为研究ADAR的功能、调控机制等后续研究提供了基础;同时,该研究揭示的ADAR与底物相互作用及催化特性为研究人员开发高效的RNA编辑工具提供了资源宝库及改进方向。
据悉,张锐教授团队的特聘副研究员宋玉龙博士及博士生杨文兵为该论文的并列第一作者,张锐教授为通讯作者。
张锐课题组研究方向为RNA组学研究和RNA疾病治疗,实验室诚聘两个方向的博士后。A. RNA生物学方向:具有细胞分子生物学背景,或者高通量测序技术开发背景,或者小鼠等动物实验模型经验背景;B. 生物信息学方向:具有较好的生物背景基础和熟悉高通量测序数据分析。
中山大学博士后在站期限为3年,因项目进展需要可申请至4年。岗位待遇:1. 基本工资年薪20万(税前)。2. 子女就读中山大学附属幼儿园和附属小学。3. 学校提供校内博士后公寓。4.协助申请广东省各类博士后人才项目。更详细的课题组介绍请见:http://lifesciences.sysu.edu.cn/teachers/professor/300
原文链接:https://doi.org/10.1038/s41594-020-0398-4参考文献
1. Nishikura, K. A-to-I editing of coding and non-coding RNAs by ADARs. Nat. Rev. Mol. Cell Biol. 17, 83–96 (2016).
2. Eisenberg, E. & Levanon, E. Y. A-to-I RNA editing - immune protector and transcriptome diversifer. Nat. Rev. Genet. 19, 473–490 (2018).
3. Liddicoat, B. J. et al. RNA editing by ADAR1 prevents MDA5 sensing of endogenous dsRNA as nonself. Science 349, 1115–1120 (2015).
4. Ishizuka, J.J. et al. Loss of ADAR1 in tumours overcomes resistance to immune checkpoint blockade. Nature 565, 43-48 (2019).
5. Reardon, S. Step aside CRISPR, RNA editing is taking off. Nature 578, 24-27 (2020), doi: 10.1038/d41586-020-00272-5
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652482548&idx=6&sn=64e53c4b171b672a84d2cf18834be940&chksm=84e23840b395b1568d479fcff2acfc1226b0a9d27ea603205df50d163a1441f92daac8c14a99&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
迄今为止效率最高的硅太阳能电池
蛋白质设施用户揭示植物组蛋白分子伴侣识别组蛋白的结构基础

NCB :组蛋白K-to-M突变助力表观遗传in vivo研究

今日科技话题:“大洋一号”船、 “向阳红10”船凯旋、“天河工程”卫星及火箭研制项目启动、有机太阳能电池转化光电率最高纪录
PNAS | 复旦大学研究揭示植物组蛋白分子伴侣识别组蛋白的结构基础
董爱武/麻锦彪组合作揭示植物组蛋白分子伴侣识别组蛋白的结构基础

陈钟课题组发现组蛋白H3替换有助于植物再生中的基因转录重编程

穿着有编号的上衣,能大大提高医护人员团队合作效率

北京大学周岳课题组揭示组蛋白H2A的单泛素化调控转录热点染色质开关的分子机制

73岁院士接种新冠重组蛋白疫苗