科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-05-07
来源:BioArt
责编 | 兮
RNA修饰是真核生物重要的转录后调控方式。其中,5-methylcytosine(m5C)修饰广泛存在于tRNA和rRNA中,然而对于mRNA上是否存在m5C修饰,学界存在很大的争议:有研究团队认为,m5C在真核生物中分布十分广泛,mRNA上存在数千以至上万个m5C位点【1】;而另外的观点认为,这些m5C是实验误差造成的,真核生物的mRNA上几乎没有真实的m5C位点。这样的争议一方面是由于mRNA在总RNA中的占比很低,因此其他类型含m5C的RNA(如tRNA)会严重干扰m5C的绝对定量;另一方面,转录组范围的m5C精确测量所依赖的重亚硫酸盐测序(Bisulfite sequencing,BS-seq)存在噪音——重亚硫酸盐无法完全转化处于二级结构区域的C碱基,因此在实验中可能存在未被转化的“漏网之鱼”被错误地识别为m5C。
近日,中山大学张锐课题组于Nature Structural & Molecular Biology上发表了题为Genome-wide identification ofmRNA 5-methylcytosine in mammals的研究,揭示了m5C修饰在哺乳动物细胞系与组织的mRNA中的精确定位和动态变化;同时表明,mRNA上的m5C据有独特的序列和结构特征。

通过数据分析,作者发现已报导的“m5C位点”有一半以上是成簇分布于同一RNA分子上。这有可能是一种类似于A-to-I RNA编辑的机制,或者是重要的假阳性特征。进一步分析表明成簇的“m5C位点”通常集中在容易形成二级结构的高GC含量区域,而二级结构会阻碍重亚硫酸盐反应并造成假阳性。通过实验,作者发现成簇的“m5C位点”对针对NSUN2的RNA干扰不敏感,也不能被m5C抗体富集,因此可能是由实验引入的假阳性。根据这些特性,作者建立了一套新颖的生物信息学计算流程过滤噪音,得以精确的定位mRNA 上m5C。在NSUN2敲除的细胞系中,大多数使用该流程获得的m5C位点都失去了甲基。这些结果说明,mRNA上的m5C位点是确实存在的,但BS-seq本身存在噪音,需要小心谨慎地去除。
作者分析发现NSUN2依赖的m5C位点倾向于拥有一个3’富G的基序(motif),通常位于在一个小颈环结构的底部。这些特征与NSUN2的tRNA底物高度一致,进一步揭示了mRNA m5C 序列和结构特征的由来。此外还有一小部分m5C位点在NSUN2敲低/敲除的细胞系中维持较高的甲基化水平。这些不依赖于NSUN2的m5C位点拥有和NSUN2依赖的位点不同的序列和结构特征。通过在HeLa细胞中逐个敲低NSUN家族成员进行BS-seq,作者发现除了NSUN2,NSUN家族其他成员不调控mRNA m5C位点,暗示除了NSUN家族成员,可能存在新的甲基转移酶参与了不依赖于NSUN2 的mRNA m5C位点的催化。
作者进一步对7个人类组织与10个小鼠组织中转录组进行测序分析,分别确认了3212与2498个高置信度位点。结果发现,在不同组织中mRNA m5C位点的数量与甲基化水平有很大差异。在人和小鼠的睾丸、肝脏、心肌和骨骼肌,mRNA上有数百个高置信度的m5C位点,而在其他的一些组织则寥寥可数。另外,这些m5C位点在人和老鼠之间并不保守,说明它们很可能是受到顺式调控(cis-regulation)的。
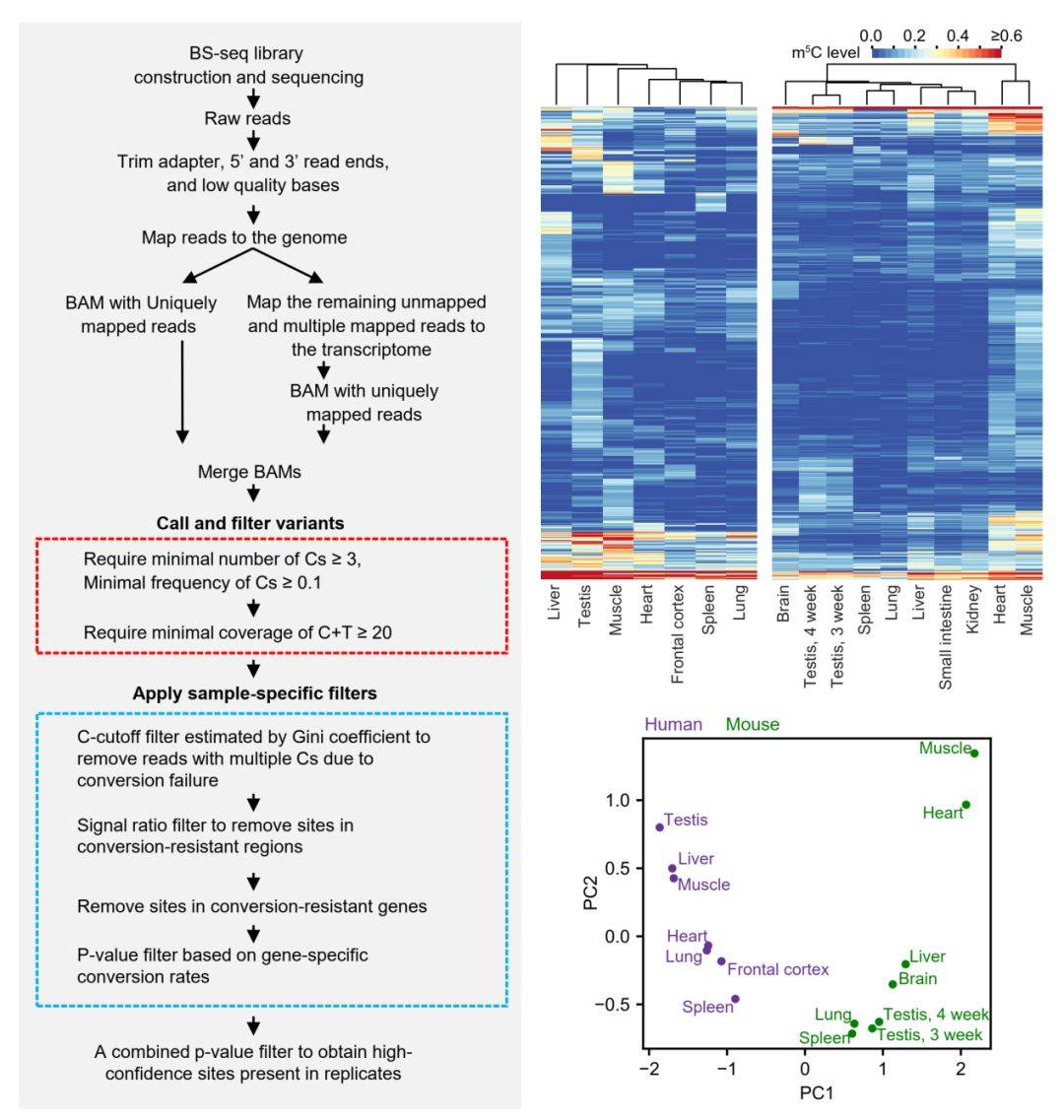
图示:mRNA m5C分析流程和动态变化规律
这项研究开发的方法为mRNA m5C的研究提供了新的工具;其构建的哺乳动物mRNA m5C 精确图谱为发现mRNA上m5C修饰的调控和功能奠定了坚实的基础。
同期的News and Views中发表了Lukas Trixl 和 Alexandra Lusser 撰写的评述文章Getting a hold on cytosinemethylation in mRNA,评价张锐课题组开发的新计算方法增加了m5C检测的精确度,进一步提供了tRNA甲基化转移酶NSUN2介导mRNA甲基化的令人信服的证据。

据悉,中山大学生命科学学院特聘副研究员黄涛、博士生陈婉颖、博士生刘健恒为该论文的并列第一作者,张锐(生命科学学院教授、孙逸仙纪念医院逸仙医学讲座教授)为通讯作者。
原文链接:
https://doi.org/10.1038/s41594-019-0218-x
制版人:小娴子
参考文献
1. Yang, X. et al. 5-methylcytosine promotesmRNA export—NSUN2 as the methyltransferase and ALYREF as an m5C reader. CellRes. 27, 606–625 (2017).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652469555&idx=3&sn=5bb6b7e20eafdf0a1d1ab0840dd152f7&chksm=84e2ea87b39563916e44d62b8fa6b4a83cee1fad8ef57af70a40a36933e1058153d3e58c3b3b&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

运动集结令 | 您身边的主动防疫微课堂(篮球:力量练习)

NSMB:桂淼、张锐合作揭示纤毛运动的机械调控结构基础

张铁锐课题组:光催化固氮的缺陷之美

张锐组揭示一个新的mRNA m5C修饰writer蛋白

马良勇、张锐合作团队鉴定早籼稻品种选育中骨干亲本核心基因组框架

张铁锐团队Nano Energy:高效光驱动费托合成制烃类化合物

中山大学张锐团队发现了新型的mRNA m5C甲基化酶

生命科学学院张锐教授团队揭示mRNA m5C的序列结构特征和动态变化规律

张锐组揭示ADAR蛋白全转录组RNA底物和决定ADAR结合和RNA编辑效率的特征

张铁锐团队: 如何提高固氮反应体系NH3检测的可靠性?