科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-07-08
来源:BioArt
染色质是指间期细胞核内由DNA、组蛋白、非组蛋白及少量RNA组成的线性复合结构,是间期细胞遗传物质存在的形式。核小体是染色质组装的基本结构单位,组蛋白八聚体构成核小体的盘状核心颗粒,由4个异二聚体组成,包括两个H2A,H2B和H3,H4。核心颗粒由145至147 bp的B型DNA盘绕组蛋白八聚体1.6-1.7圈,组蛋白H1在核心颗粒外结合额外20bp DNA,锁住核小体DNA的进出端,起稳定核小体的作用,这种结构形式使得只留有小部分的核小体DNA可以被结合因子利用【1,2】。
紫外线辐射造成的DNA损伤是常见的一种损伤形式,紫外线(UV)会诱导暴露在其下的DNA形成嘧啶二聚体,例如6-4光产物(6-4 PPs,6-4 pyrimidine-pyrimidone photoproducts)和环丁烷-嘧啶二聚体(CPDs,cyclobutane-pyrimidine dimers)。染色质致密的结构状态存在会阻碍其上的损伤DNA的修复 【3】。
核苷酸切除修复(NER,nucleotide excision repair)是修复紫外线引起的DNA损伤的主要方式之一,它包括执行全基因组修复的全基因组核苷酸切除修复(GG-NER,global genome nucleotide excision repair)和主要发生在基因转录过程中的快速修复方式转录偶联核苷酸切除修复(TC-NER,transcription-coupled nucleotide excision repair)。在GG-NER途径中,主要是由蛋白复合物XPC-RAD23-CETN2和UV-DDB(UV-damaged DNA-binding protein)检测到UV损伤。UV-DDB由蛋白DDB1和DDB2组成,与DDB1相关的cullin-RING E3泛素连接酶CUL4A(或CUL4B)协同工作。在识别UV损伤之后,泛素连接酶被激活,损伤位点附近的蛋白质,包括组蛋白均被泛素化。研究表明,UV-DDB结合损伤位点要先于染色质重塑相关蛋白机器和XPC,并且DDB2参与识别的DNA损伤位点与损伤位置是否在核小体内或连接DNA(linker DNA)上无关【4】。早在1988年,G. Chu和E. Chang在Science上就已报道了DDB2基因在着色性干皮病基因互补E组的患者中存在着特异性的突变【5】,这类患者体内存在罕见的先天性DNA修复障碍,导致其对紫外线极度敏感,其皮肤癌的发病率增加近1万倍【6】。
尽管UV-DDB如何识别UV诱导损伤的双链DNA并与之结合的结构基础已经被很好的解析,然而,由于核小体特殊“访问限制”(access restrictions )的存在,目前我们还不清楚UV-DDB是如何识别核小体中的紫外损伤区域。无论如何,识别并结合DNA这一过程不仅是DNA损伤修复的基础,也与所有与染色质基因组DNA结合的蛋白质(例如转录因子)休戚相关。因此,阐明UV-DDB如何识别核小体DNA上不同位置的UV损伤至关重要。
近日,Nature杂志在线发表了由瑞士弗里德里希·米舍尔生物医学研究所(Friedrich Miescher Institute for Biomedical Research)Nicolas H. Thomä团队题为DNA damage detection in nucleosomes involves DNA register shifting 的长文,报道了UV-DDB与在不同位置存在6-4光产物或是类DNA损伤的核小体结合的冷冻电镜结构,发现UV-DDB在不影响核小体整体结构的情况下,会结合在UV损伤核小体的溶剂暴露面的小沟处。而在面对组蛋白核心面的隐匿性突变中,UV-DDB会改变核小体的主要翻译域,并选择性地将突变位置固定在可接近的暴露位置。这一工作为发生在核小体上的DNA损伤检测提供了有力的结构支持。

为测定UV-DDB在含嘧啶二聚体的核小体复合物中的结构,研究人员首先构建了由145-bp 人类α卫星DNA和组蛋白核心蛋白H2A,H2B,H3.1和H4组成的核小体八聚体核心成分(NCPs,nucleosome core particles)。其中,正向DNA链上包含了一个6-4PP的单一突变或者是包括两个连续的四氢呋喃核苷酸碱基位点(THF2,tetrahydrofuran-nucleotide abasic sites,是UV-DDB的配体)。(图1)
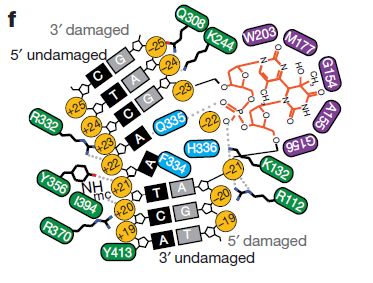
图1 . NCP6-4PP-UV-DDB复合体示意图
为了使NCP6-4pp与UV-DDB之间的亲和性最大化,研究人员设计的核小体上的6-4pp位置在二分体轴-22/-23bp位置。利用单颗粒冷冻电镜(cryo-EM)技术,研究人员重建得到了分辨率为4.3Å的NCP6-4pp-UV-DDB复合体结构。发现主要的接触位于DDB2螺旋和核糖体DNA超螺旋位置(SHLs, superhelix locations)-2和-3处的小沟上。DDB2相对于平行于核小体盘状核心颗粒的平面的角度约为60°,而DDB1远离核小体核心并且不与核小体相互作用。进一步地解析DDB2结构,发现DDB2中带正电荷残基的侧链与DNA主链连接超过6 bp(-20bp到-25bp),并与Gln308,Tyr356,Ile394和Tyr413形成氢键和疏水堆积相互作用。DDB2结合损伤位点周围的DNA小沟并插入β-hairpin loop组成Phe334-Gln335-His336残基是其识别机制的一部分。β-hairpin的插入需要6-4PP被挤压形成extra-helical构象,而DDB2残基Gln335和His336稳定挤压后的损伤位点对面的两个孤立碱基。(图2)
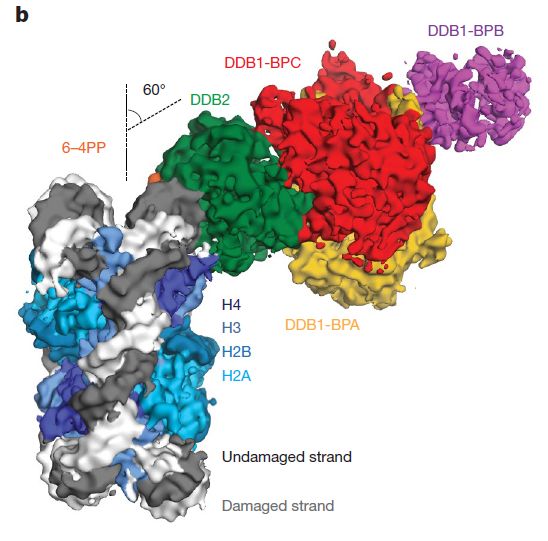
图2. NCP6-4PP-UV-DDB复合体结构(分辨率为4.3Å)
研究人员通过比较与含有6-4PP的核小体结合的UV-DDB和与含有6-4PP的DNA双链结合的UV-DDB的结构,发现两者之间的大部分UV-DDB-DBA的接触结构是保守的。此外,紫外损伤的核小体的识别机制与分离出来的含有嘧啶二聚体的DNA双链的识别机制并无不同。
尽管在构建的核小体结构中DDB1 的B-domain是部分无序的,研究人员根据cryo-EM map对DDB1 B-domain blades 1,2,3,6,7的特征进行分析并确定它们的区域坐标,进而得到了CRL4DDB2 E3连接酶复合体的模型。连接酶在核小体核心周围旋转这一结构特征也解释了之前报道的核小体附近组蛋白尾和不同的核心组蛋白泛素化这一现象。
接下来,研究人员检测了UV-DDB是否可以与暴露在小沟-22/-23之外的受损位点接触。研究人员模拟了UV-DDB与覆盖了SHL-1.5至SHL-2.5的核小体DNA片段的结合,并计算了UV-DDB与核小体之间空间冲突程度。碰撞分数沿超螺旋区段的总体分布为U型,即UV-DDB最佳的结合位点处于溶剂暴露面(solvent-facing)的小沟处,这也与cryo-EM结构所述一致。
紧接着,研究人员利用荧光偏振竞争分析了在SHL-2前后不同位点UV-DDB的THF2损伤情况,发现亲和度最高的位点是0点,同时也是预测的最具可访问性的位置。相反地,UV-DDB对位于自由的145-bp DNA双链上的THF2位点的结合亲和性没有位点相对差异。因此,核小体使DDB2更难以接近向内的损伤位点。
那么UV-DDB是如何适应这些冲突并实现向内面(inward-facing)的损伤位点识别的呢?
研究人员通过构建NCP-3位上的THF2突变与UV-DDB形成复合体UV-DDB (NCPTHF2(-3)-UV-DDB) ,发现DDB2与损伤的核小体结合而不与核心组蛋白或者是两股DNA螺旋发生空间冲突。并且,UV-DDB的结合会促进存在3-bp 核小体DNA储存器(人α卫星DNA序列)的位移。进一步的,利用DNase I消化存在和不存在UV-DDB情况下的含损伤位点的NCPs,通过下一代测序,研究人员分析并确定了样本中所有的核小体储存器。确定了在UV-DDB存在的情况下,存在一种重新定位机制,将主要闭塞的面向组蛋白面的损伤位点移动至与DDB2识别兼容的旋转位置中。此外,研究人员还发现UV-DDB稳定的核小体DNA储存器精确定位和DNA双向运动依赖于损伤位置。
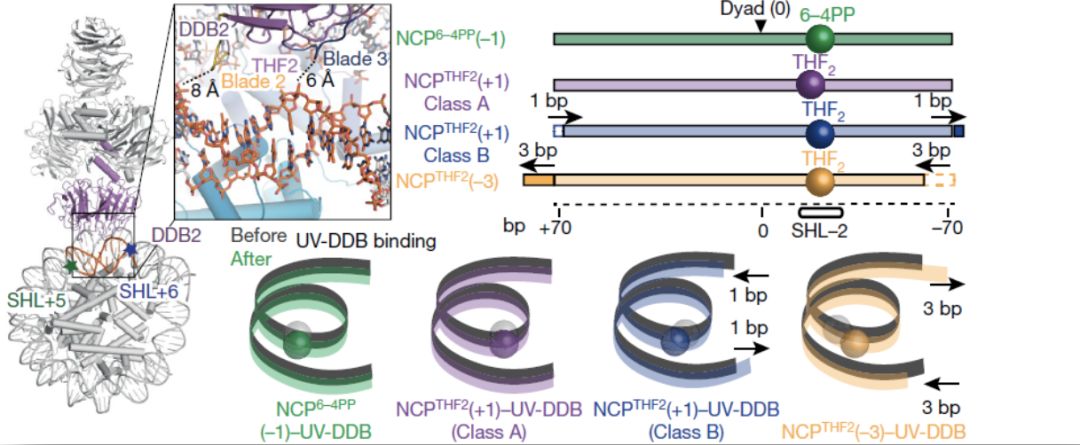
图3. UV-DDB结合损伤DNA
最后,研究人员通过比较UV-DDB对不同核小体(DNA的Watson链和Crick链上不同位置的突变,NCP6-4PP(-1:Watson)-UV-DDB, NCPTHF2(-1:Crick)-UV-DDB)的结合能力发现了局部区域的环境以及损伤位点的空间构象的可入性还有核小体-DNA接触强度决定了UV-DBD对染色质损伤区域的亲和力。
总的来说,本研究为蛋白机器如何有效识别染色质的紫外损伤提出了一个结构和机制上的框架,并进一步阐述了DNA结合蛋白是如何获得许可结合核小体DNA。通过利用核小体-register的动力学特性,UV-DDB双向作用于DNA储存器。同时UV-DDB检测核小体上的损伤位置不依赖于ATP驱动的染色质重组机器,也不需要八聚体的去组装或核小体DNA环化。取而代之的,是UV-DDB克服内在作用逐步定位于人α-卫星DNA重复序列上并且滑动,识别损伤位点。
原文链接:
https://doi.org/10.1038/s41586-019-1259-3
参考文献
1. McGinty, R. K. & Tan, S. Nucleosome structure and function. Chem. Rev. 115, 2255–2273 (2015).
2. Luger, K., Mäder, A. W., Richmond, R. K., Sargent, D. F. & Richmond, T. J. Crystal structure of the nucleosome core particle at 2.8 Å resolution. Nature 389, 251–260 (1997).
3. Hanawalt, P. C. & Spivak, G. Transcription-coupled DNA repair: two decades of progress and surprises. Nat. Rev. Mol. Cell Biol. 9, 958–970 (2008).
4. Fei, J. et al. Regulation of nucleotide excision repair by UV-DDB: prioritization of damage recognition to internucleosomal DNA. PLoS Biol. 9, e1001183 (2011).
5. Chu, G. & Chang, E. Xeroderma pigmentosum group E cells lack a nuclear factor that binds to damaged DNA. Science 242, 564–567 (1988).
6. Lehmann, A. R., McGibbon, D. & Stefanini, M. Xeroderma pigmentosum. Orphanet J. Rare Dis. 6, 70 (2011).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652472161&idx=1&sn=92e28e5356ec3c806fd8166583a9217a&chksm=84e210d5b39599c39f9b1577602d9762d0fba483f7ed0235c5e20afc6b02123003f8199cb637&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
【大师讲堂】神奇的DNA损伤修复

Nature:黄晶课题组揭示核小体结构对组蛋白修饰酶复合物的酶活调控及其分子机制
非对称聚合物材料可制造灵活多变的软机器人

揭示核小体结构对组蛋白修饰酶复合物的酶活调控及其分子机制
DNA修复之谜
李祥/阮永怡/黄永瀚合作揭示组蛋白戊二酰化对核小体动态结构的影响



研究揭示组蛋白H2A泛素化修饰对核小体的调控机制
Nature : 刘扬/周钶达等揭示组蛋白分子伴侣FACT与亚核小体作用的机制
王艳丽:做研究就像破案

核小体的编辑具有温度敏感性--DNA序列在组蛋白变体的组装过程中起重要作用