科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-09-15
来源:BioArt
编者按
从冈崎令治夫妇发表关于“冈崎片段”的里程碑式文章到现在,时间已经过去了五十年,但DNA复制过程中前导链的连续性问题一直没有得到很好的解释。
2019年1月22日,来自伊利诺伊大学香槟分校的Andrei Kuzminov团队在PNAS上发表了题为Near-continuously synthesized leading strands in Escherichia coli are broken by ribonucleotide excision的研究文章,对DNA复制过程中前导链的不连续性问题做了详细的阐述。而源于这项工作的重要性,PNAS特邀华盛顿大学医学院的教授Peter M. Burgers对本文工作进行了详细的点评。Peter M. Burgers教授的研究方向是DNA复制和DNA损伤反应,在DNA复制领域发表了一系列的文章,以下是Peter M. Burgers教授的评论内容。
原文丨Peter M. Burgers
编译丨秋枫
责编丨迦溆

由于双链DNA具有反向平行结构,以及众所周知的从5’向3’运行的DNA聚合酶,因此,双链DNA的复制需要在相反的方向上进行。前导链的合成与复制叉同向,而后随链的合成则与之反向。在冈崎令治和冈崎恒子以及他们的团队于1968年发表在PNAS上的文章【1】中提出了一种假设,DNA的后随链以小片段的不连续形式进行复制,最后小的片段结合形成连续的成熟复制链。当年,在一个关于当时分子生物学的综述【2】里,这种不连续的复制片段被命名为“冈崎片段”,从那以后,这个名字一直被沿用至今。冈崎夫妇的研究确立了关于复制叉半不连续复制的教科书级模式,即前导链的连续复制和后随链的非连续复制。然而,他们最初的实验结果并不符合这种模式,他们的研究表明,所有新合成的DNA都是小的片段。难道,前导链的合成也是不连续的,就像Fig.1 C描述的那样?在冈崎夫妇这篇里程碑式的文章发表50年之后,这个问题终于被Cronan等【3】解决了,他们的研究表明,前导链的合成确实是连续的,但是这种连续的链被核苷酸切除修复(ribonucleotide excision repair, RER)【4,5】打断成了小的片段。RER移除了复制过程中被DNA聚合酶错误添加的核苷酸(Fig.2A)。
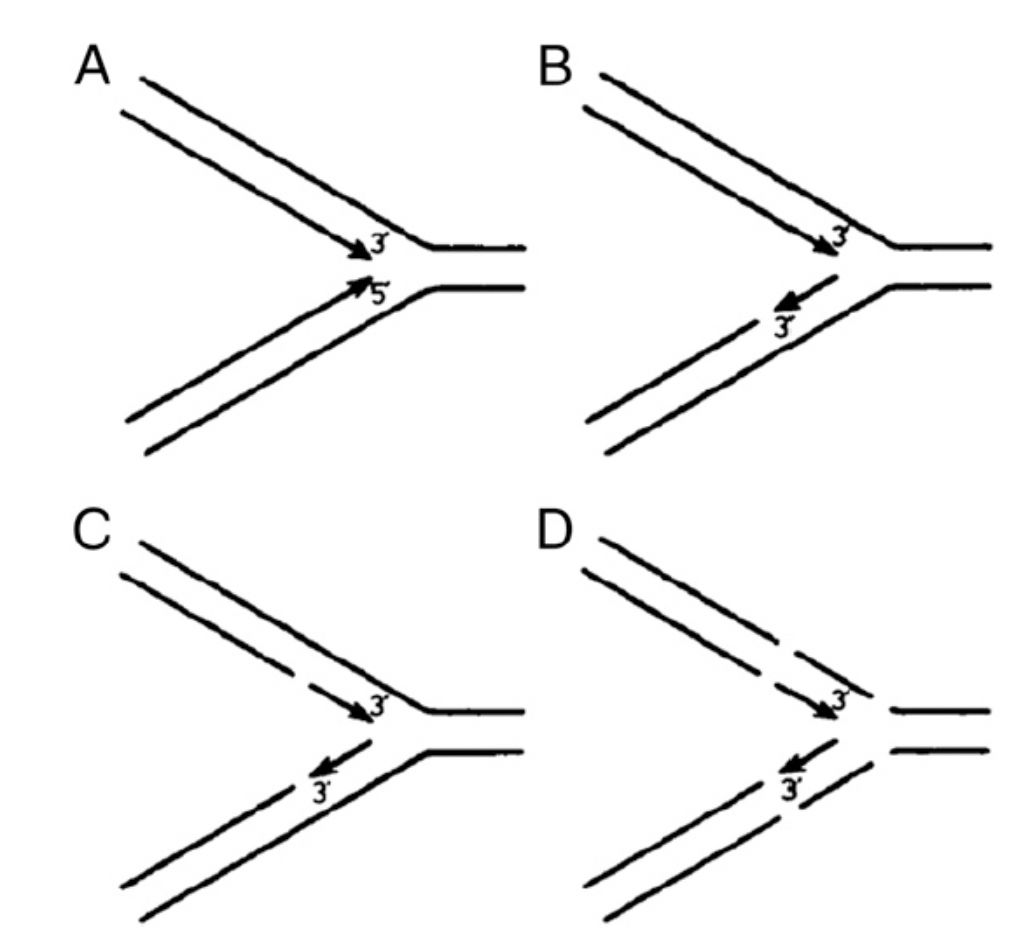
Fig.1 DNA复制可能的结构方式
为了检测最早的后随链片段,保证在它们被合成连续的成熟链之前完成检测,冈崎团队在大肠杆菌里采用了脉冲标记技术,用3H标记胸腺嘧啶的脉冲只维持5秒。因为DNA的复制速率大约为500到1000 nt/s,而一个冈崎片段的平均长度一般为1到2 kb,所以只需要几秒钟的时间一个冈崎片段便能被合成。脉冲标记的DNA经过变性后,用于碱性蔗糖梯度分离。令人惊讶的是,虽然同时进行的生化研究表明,前导链的合成是以连续的方式进行的【6】,但实际上,所有标记的DNA分级都在级别的最上边,在DNA的长度上大约为1到2 kb。仅仅有很小的一部分标记的DNA长度在10 kb以上,结果与Fig.2B中的黑色曲线类似。很显然,这个结果支持了Fig.1C的模式而不是理想中的Fig.1B的DNA半不连续复制模式。而且,这种奇怪的结果并不仅仅局限于大肠杆菌,在革兰氏阳性菌枯草芽孢杆菌和T4噬菌体中也存在着相同的现象【7】。
接下来的几年中,冈崎团队在研究中又取得了一些新的进展,加深了对冈崎片段的认识。他们的结果表明,在额外添加冷胸苷的情况下,被脉冲标记的小片段能够连接形成大的片段,表明这些小的片段确实是染色质DNA合成的中间形式。他们发现,冈崎片段的合成是由一个短的RNA引物起始的【8】。他们的研究还表明,DNA合成的方向在体外和体内是相同的,都是由5’向3’运行,而RNA引物也确实存在于冈崎片段的5’端。然而,尽管取得一系列进展,但最初合成的DNA是小片段的事实仍旧存在,前导链复制的真实情况依然是最需要研究清楚的问题。在冈崎令治于1975年英年早逝之后,冈崎恒子于名古屋大学中将这项由她和她丈夫起始的开创性研究延续了下去。她的团队分别在原核生物和真核生物钟鉴定了RNA引物的结构【9】。冈崎恒子近些年关于复制中间形式的研究可在ref.10中查询。
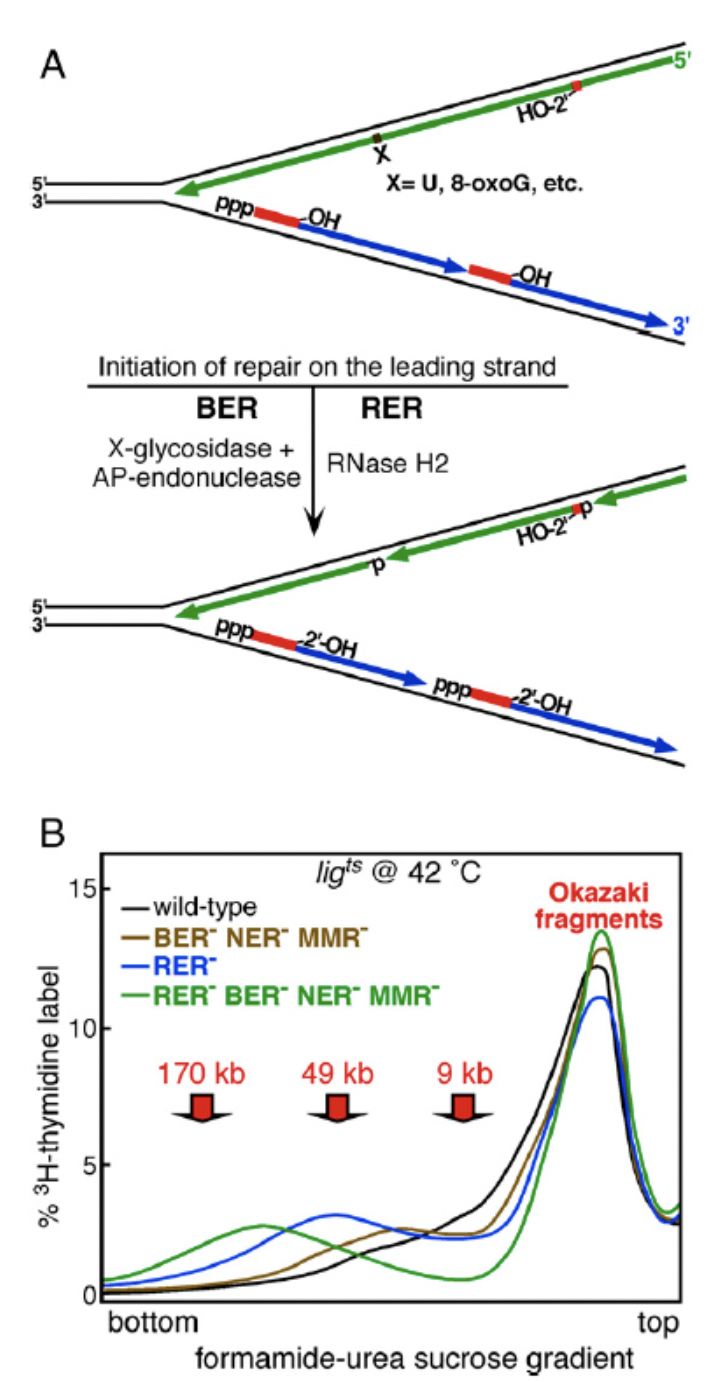
Fig.2 (A) DNA的切除修复模式;(B) 甲酰胺-尿素-蔗糖分级后DNA的大小分布情况
研究人员最初发现一些小片段有可能源于DNA修复,是在用脉冲标记研究大肠杆菌中冈崎片段突变(sof, short Okazaki fragment) 的实验中观察到的【11】。在sof的突变体中,能够审查dNTP池并将错误合成的dUTP水解的酶dUTPase是缺陷的。dUTP这种类似物能够在复制过程中非常容易的插入到DNA的结构中,但是尿嘧啶碱基会在接下来的过程中被尿嘧啶-DNA-糖苷酶切除掉,这是碱基切除修复(base excision repair, BER)的基本功能。而在sof突变体中,高水平的dUTP便导致了高频率的dUMP错误插入以及高频率的BER,最终产生的结果便是新和成的DNA被严重片段化。然而,在野生型的细胞中,很明显尿嘧啶切除并不是DNA复制过程中产生小片段的原因。但尽管如此,这个实验却让我们想到DNA修复有可能是前导链形成小的片段的原因。那么,究竟是哪种DNA修复方式呢?实验人员设计了所有碱基切除修复(BER)、核苷酸切除修复(NER)和错配修复(mismatch repair, MMR)的突变体,但仍然没有得到DNA半不连续复制模式中的双峰分布结果【12】。
其实,在冈崎团队发表他们的重要成果之前,科学家一致认为DNA聚合酶能够利用三磷酸核糖核苷(rNTP)作为底物,虽然这种情况很少见【13】。而对基因组核糖核苷酸性质和丰度研究的关键成果来自于酵母菌的研究。处于复制状态的DNA聚合酶能够粗略的辨别dNTP和rNTP,辨别比率大约为104到105:1。乍一看,如此低的错误比例不应产生很严重的结果才对。然而,四种rNTPs在细胞内的浓度比相对应于dNTPs的浓度高出约30到200倍。因此,按照一种简单的竞争机制,当rNTPs和dNTPs都处于他们的生理浓度时,更高频率的复制错误会产生,比例大约为每一千个碱基对应一个三磷酸核糖核苷【14】。这些基因组中的三磷酸核糖核苷通过RER途径被切除了,作用机理是在三磷酸核糖核苷的5’位置被RNA酶H2(RNase H2)催化产生一个切口(Fig.2A)。核苷酸切除修复途径(RER)特异性的使用RNA酶H2(在大肠杆菌中为RnhB),因为RNA酶H1不能切除基因组中的单个核苷酸。在大肠杆菌中,dNTP池并不像酵母菌中那么小,尽管如此,dATP的浓度也只是ATP浓度的二十分之一【15】。如此以来,三磷酸核糖核苷的错误插入很可能就是半个世纪以来前导链片段问题的来源了。
为了验证他们的分析,Cronan团队采用了一种包含温度敏感连接酶的菌株。在这个菌株中,当温度由28上升到42°C时,DNA连接酶的活性会急剧消失,这样便能阻止冈崎片段以及切除修复事件中的连接反应。另外,他们对最初冈崎团队采用的实验步骤进行了一些重要的改进。因为预测到当大肠杆菌中rnhB突变导致RER失效后,三磷酸核苷将留在基因组中,因此蔗糖梯度分级将无法在大小上区分开最初合成的单链DNA。而RNA对碱性是敏感的,因此如果在碱性条件下离心,DNA将在三磷酸核苷插入的位置被打断为片段。因此,他们转而采用甲酰胺-尿素-蔗糖的分级方法,并且在pH中性的条件下进行实验,这样能够使变性的DNA得到相似的分离效果。通过这种改进,他们首先研究了BER,NER和MMR的失活突变中标记DNA的大小分布。与野生型相比,和冈崎片段大小相似的产物的高分子量峰肩向左边稍微偏移了一些,说明新合成的DNA发生了一些修复事件。然而,比50kb大的产物却消失了。值得注意的是,rnhB突变株的产物出现了双峰的分布情况,高分子量产物集中在50kb左右(Fig.2B,蓝色曲线)。最后,当所有的修复途径都被失活时,一个粗略的高分子量分布峰出现了,它的大小大约是80kb(Fig.2B,绿色曲线)。重要的是,超过90%的小的片段都属于后随链,而超过90%的大片段DNA都属于前导链。这些数据与冈崎团队早年的研究结果是相符的【16】,其中DNA聚合酶I的突变能够导致所有小片段的连接失效,因为在大肠杆菌中DNA聚合酶I既是RER需要的,也是冈崎片段的连接成熟需要的。
Cronan团队的新研究表明,三磷酸核糖核苷的插入以及被RER切除在DNA合成中频繁发生。为了评估这一事件的频率,Cronan团队测量了在rnhB突变株中传播的含有三磷酸核糖核苷质粒的数量。假设三磷酸核糖核苷的插入是完全随机的,Cronan团队采用泊松计算式对由RNase H2在分离到的质粒上打断的缺口数量进行统计分析,得到的错误插入频率约为每8到9kb中有一个三磷酸核糖核苷,或者每个基因组中有500个。RER在细菌中是应用最频繁的DNA修复途径,与其在真核细胞中的应用频率在一个数量级上。
当前的研究已经证实,前导链的合成大体上是连续的,现在的问题是它是怎样实现这种连续的?复制叉会因为各种原因而停止前进,细胞也有多种机制让复制叉重新启动【17】。其中一些重启机制是重组或者复制叉重塑后利用3’端重启,但是RNA引物也可以作为一种重启复制的机制。复制重启后合成的DNA将是片段化的,而且出现在前导链上。通过对多种切除修复途径突变株(RER-BER-NER-MMR-)中前导链的大小分布的研究,Cronan团队证实,前导链的合成确实不是完全的连续的。而这种不连续到底是源于某一种修复途径,或者源于复制叉的重新启动,又或者是源于前导链上DNA引物酶的随机变化边需要将来更深入的研究了。
参考文献
1 Okazaki R, Okazaki T, Sakabe K, Sugimoto K, Sugino A (1968) Mechanism of DNA chain growth. I. Possible discontinuity and unusual secondary structure of newly synthesized chains. Proc Natl Acad Sci USA 59:598–605.
2 Anonymous (1968) Molecular biology comes of age. Nature 219:825–829.
3 Cronan GE, Kouzminova EA, Kuzminov A (2019) Near-continuously synthesized leading strands in Escherichia coli are broken by ribonucleotide excision. Proc Natl Acad Sci USA 116:1251–1260.
4 Sparks JL, et al. (2012) RNase H2-initiated ribonucleotide excision repair. Mol Cell 47:980–986.
5 Vaisman A, et al. (2014) Investigating the mechanisms of ribonucleotide excision repair in Escherichia coli. Mutat Res 761:21–33.
6 Goulian M, Kornberg A, Sinsheimer RL (1967) Enzymatic synthesis of DNA, XXIV. Synthesis of infectious phage phi-X174 DNA. Proc Natl Acad Sci USA 58:2321–2328.
7 Okazaki R, et al. (1968) In vivo mechanism of DNA chain growth. Cold Spring Harb Symp Quant Biol 33:129–143.
8 Sugino A, Hirose S, Okazaki R (1972) RNA-linked nascent DNA fragments in Escherichia coli. Proc Natl Acad Sci USA 69:1863–1867.
9 Kitani T, Yoda K, Okazaki T (1984) Discontinuous DNA replication of Drosophila melanogaster is primed by octaribonucleotide primer. Mol Cell Biol 4:1591–1596.
10 Okazaki T (2017) Days weaving the lagging strand synthesis of DNA—A personal recollection of the discovery of Okazaki fragments and studies on discontinuous replication mechanism. Proc Jpn Acad, Ser B, Phys Biol Sci 93:322–338.
11 Tye BK, Chien J, Lehman IR, Duncan BK, Warner HR (1978) Uracil incorporation: A source of pulse-labeled DNA fragments in the replication of the Escherichia coli chromosome. Proc Natl Acad Sci USA 75:233–237.
12 Amado L, Kuzminov A (2013) Low-molecular-weight DNA replication intermediates in Escherichia coli: Mechanism of formation and strand specificity. J Mol Biol 425:4177–4191.
13 Berg P, Fancher H, Chamberlin M (1963) The synthesis of mixed polynucleotides containing ribo-and deoxyribonucleotides by purified preparations of DNA polymerase from Escherichia coli. Informational Macromolecules, eds Vogel HJ, Bryson V, Lampen JO (Academic Press, New York), pp 467–483.
14 Nick McElhinny SA, et al. (2010) Abundant ribonucleotide incorporation into DNA by yeast replicative polymerases. Proc Natl Acad Sci USA 107:4949–4954.
15 Buckstein MH, He J, Rubin H (2008) Characterization of nucleotide pools as a function of physiological state in Escherichia coli. J Bacteriol 190:718–726.
16 Okazaki R, Arisawa M, Sugino A (1971) Slow joining of newly replicated DNA chains in DNA polymerase I-deficient Escherichia coli mutants. Proc Natl Acad Sci USA 68:2954–2957.
17 Marians KJ (2018) Lesion bypass and the reactivation of stalled replication forks. Annu Rev Biochem 87:217–238.
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652475294&idx=1&sn=8e8f1faa9e77da31b9bfcf6f391a99a9&chksm=84e21c2ab395953c6d90e935eb94b4e3828d208726f314a7b92301b15e78bf8bdf7f1d04dd94&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

新研究揭开人类头发曲直的秘密
【大师讲堂】解读生命密码:DNA的复制历程
演化:“最奇怪动物”身份揭秘
有关如何读取和复制DNA的新研究

《Cell》更强劲的DNA修复=长寿
非对称聚合物材料可制造灵活多变的软机器人

【大师讲堂】解读生命密码:DNA的复制历程

【大师讲堂】解读生命密码:DNA的复制历程

自然中不存在的蛋白质也能人造了!撼动材料医药界
王艳丽:做研究就像破案