科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-10-18
来源:Deep Omics
原标题:Nature Methods文章:基于基因互作网络的突变图谱非负矩阵分解模型(NBS)揭示肿瘤亚型
文章题目:
Network-based stratification of tumour mutations
发表时间:2013年9月
期刊名称:Nature Methods
PMID号:24037242
关键词: 基因互作网络; 非负矩阵分解(NMF); 肿瘤突变图谱; 亚型分类; The Cancer Genome Atlas (TCGA);
本文提出了一种基于网络的分类模型(Network-Based Stratification,NBS),该方法首先利用基因互作网络数据库对癌症病人的肿瘤突变图谱进行去噪与缺失值填充,然后将具有相似突变的患者聚集在一起,由此来将癌症病人分为不同的亚型。研究人员展示了NBS模型能够很好地分别对TCGA卵巢癌,子宫癌和肺癌进行亚型分类,并发现这些亚型与临床信息高度关联,例如患者预后,治疗反应,肿瘤病理类型等信息。

Nat Methods. 2013 Nov;10(11):1108-15. doi: 10.1038/nmeth.2651.
【NBS模型介绍】
NBS是一种非负矩阵分解(NMF)方法,它的输入是一个肿瘤队列的突变图谱矩阵(每行为基因,每列为病人)。给定一个分类数(k),NBS模型会利用基因互作网络和NMF把病人分为k个不同的亚型(图1a)。相比于传统NMF分类方法,NBS利用基因互作网络进行了以下两点创新:
① 利用网络传播算法对病人的突变谱进行去噪、缺失值填充处理(图1b);
② 在训练目标函数时加入了基因互作网络的约束(图1c)。
由于NBS模型中引入基因互作网络的先验知识,大大地提高后续无监督聚类(如一致性聚类(consensus clustering),图1d)的准确性。
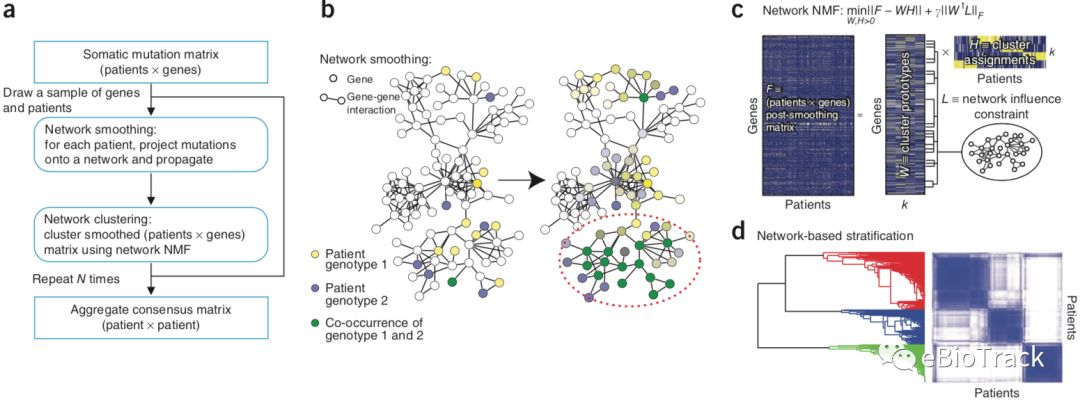
图1,即文章的Figure 1. NBS算法概况。
【NBS在模拟数据上的表现】
为了验证NBS模型的效果,研究人员首先模拟了具有显著亚型特征的基因互作网络和突变表达谱(图2a)。然后评估了模拟数据在以下四种模型里的表现:
① 基于显著特征基因网络的NBS(NBS);
② 一致性聚类;
③ 基于无显著特征基因网络的NBS(permuted NBS);
④ 将NBS中的NMF换为层次聚类。
“基于显著特征基因网络的NBS”的亚型聚类准确度远远大于其他三种模型设置(图2b)。所以,“显著特征基因网络”和“NMF”这两个步骤在NBS模型中缺一不可。然后,研究人员调查了“基因突变频率”和“基因互作范围”对模型的影响。结果表明,在高频突变和小范围基因互作的情况下,“一致性聚类”(图2d)和“permuted NBS”(图2e)两个模型表现较差,而NBS模型依然能够解出正确的亚型(图2c)。
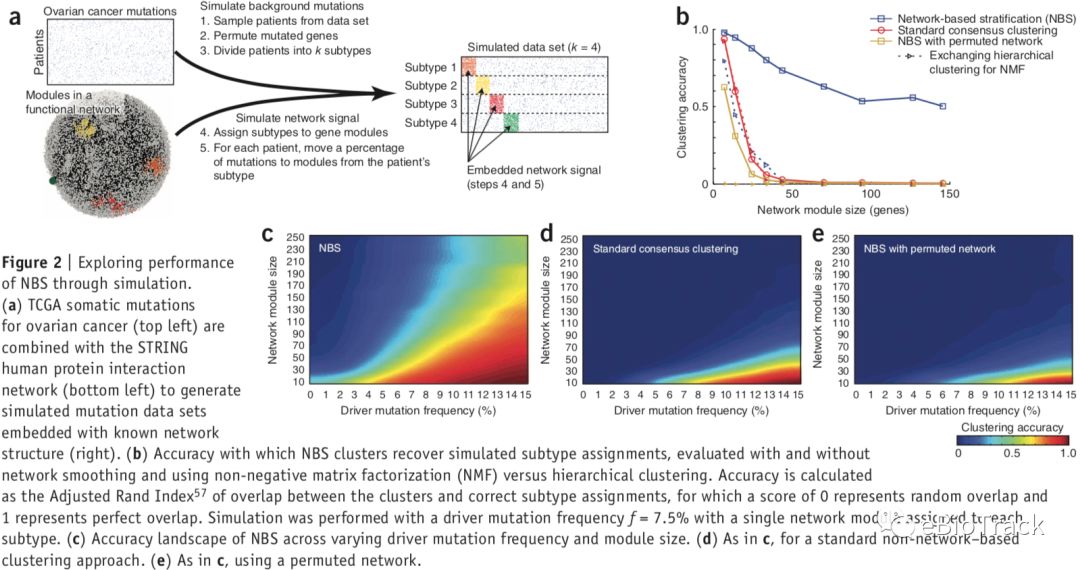
图2,即文章的Figure 2. NBS在模拟数据上的表现。
【NBS在TCGA数据上的表现】
研究人员将NBS应用到TCGA突变图谱数据上(全外显子组测序),分别对子宫癌,卵巢癌和肺癌患者进行分型。相较于“一致性聚类”,NBS能清晰解出每种癌症的亚型结构(图3a为子宫癌;卵巢癌和肺癌结果请查看论文附图3a和4a)。 研究人员调查了不同模型解出的亚型分类与临床信息(例如患者预后,治疗反应,肿瘤病理类型)之间的关联。这些模型是:
① 基于显著特征基因网络的NBS(NBS);
② 一致性聚类;
③ 基于无显著特征基因网络的NBS(permuted NBS);
结果表明,三组数据上,“基于显著特征基因网络的NBS”模型都解出了明显优于其他两种模型的亚型:其解出的子宫癌的亚型(图3b)和肿瘤病理类型(图3c)显示出强关联;解出的卵巢癌亚型(k=4)显示出与预后的强关联(图3d-e);解出的肺癌亚型(k=6)显示出与预后的强关联(图3f-g)。
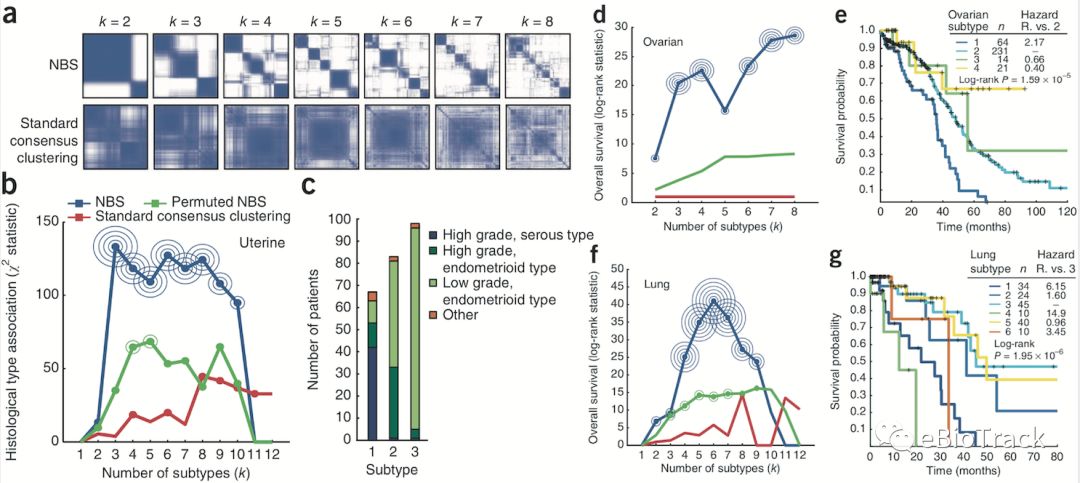
图3,即文章的Figure 3. NBS在TCGA数据上的表现。各小图位置重构以方便展示。
研究人员将基于基因突变图谱(经基因互作网络处理)的NBS亚型与从其他数据(未经基因互作网络处理)得到的亚型进行了比较。其他数据包括,拷贝数变异,甲基化,mRNA表达,microRNA表达和蛋白质谱。对于卵巢癌,所有其他数据类型具有较差的预测存活率的能力(详见论文Fig4a-b)。在肺癌中,NBS亚型和基于RNA-seq的亚型具有良好的预测能力(详见论文Fig4c-d),而其余数据类型不能很好地预测存活率。在子宫癌中,所有数据类型的亚型都对病理学分类具有高度预测性(详见论文Fig4e),并且与NBS亚型具有非常高的重叠(详见论文Fig4f)。
【生物学探讨】
研究人员发现FGF通路在卵巢肿瘤亚型1中占主导地位(详见论文Fig5),FGF通路涉及肿瘤增殖和血管生成,已有文献显示FGF通路的表达增加与卵巢癌预后差相关,抑制FGFR1和FGFR2的基因表达可以增加癌细胞对治疗药物(如顺铂,Cisplatin,CDDP)的敏感性。
另一个有趣的发现是长片段基因在癌症基因网络中的富集(详见论文Fig7)。对于卵巢肿瘤亚型2来说,子网络176个基因中有12个基因的长度在前2%(例如TTN,已知最长的编码基因)。虽然癌症体细胞突变目录(COSMIC)和Vogelstein名单也表现出长基因富集,关于这些基因在癌症中的作用仍然存在争议。一方面,长基因高度突变的原因可能不是因为它们是癌症的驱动基因,而仅仅是因为它们是一个更大的“目标”,有更多的突变概率。另一方面,没有明确的证据表明长基因的突变对肿瘤进展没有贡献。研究人员的分析证明了在癌症研究中长基因不应该被忽视。
另外,研究人员发现尽管同义突变对子宫癌和肺癌的分子分型作用不大,但同义突变对卵巢癌分子分型具有一定的预测能力(详见论文Fig7)。已经有一些研究表明,同义突变确实可能在癌症进展中发挥因果作用。研究人员表明,需要进一步研究来了解同义突变是否以及如何在卵巢癌中真正起作用。
【我们的思考】
和传统的NMF亚型分类方法相比,本文进行了以下两点创新:① 利用网络传播算法对基因突变谱进行去噪、缺失值填充处理;② 在训练目标函数时加入了基因互作网络的约束。基因互作网络的引入,大大提高了后续无监督聚类的准确度。
如果有病人的临床信息(如各项检测指标),我们也可以通过高斯核函数计算出病人之间的相似度网络,加入NBS算法的目标函数中以协助分类,这样可能得到与预后更加相关的亚型。
研究仅将基因互作网络结合突变图谱,其实NBS的思想可以运用到任何行是基因的图谱中,例如基因表达谱,基因甲基化谱,甚至是多组学组成的混合矩阵。
来源:gh_cc101106ed81 Deep Omics
原文链接:http://mp.weixin.qq.com/s?__biz=MzI2NDg2MjgyOA==&mid=2247483650&idx=1&sn=8c39aafbcb9f26ac9de7768354790ab3&chksm=eaa76c01ddd0e517d2b04306553e0138a0149998b660da15fd6414ed0f451b5b08525f6e4b96&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

人工智能专家王笛为你详解基于矩阵分解的哈希跨媒体检索方法
机器如何区分芒果的酸甜?分类和聚类需了解
模糊目标聚类
动态聚类法

人工智能、深度学习、神经网络、大数据备忘录

【速览】ICCV2019 | 基于深度综合相关性挖掘的图像聚类算法
无监督量子聚类算法
系统聚类法

全新给药方式可降低癌症复发率

发光纳米颗粒可以更快更精准地检测癌症