科技工作者之家
科界APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-01-31
来源:植物科学最前沿
原标题:PBJ:安徽省农科院/安徽农大合作通过高密度碱基编辑技术进行定向进化获得水稻ACCase抗性突变体
近日, 安徽省农业科学院水稻研究所魏鹏程组与安徽农业大学农学院吴德祥组合作在国际著名期刊Plant Biotechnology Journal (即时影响因子8.4)上发表了题为“A CRISPR-Cas9-mediated domain-specific base-editing screen enables functional assessment of ACCase variants in rice”的通讯短文。该研究通过构建碱基编辑编辑文库,实现植物体内重要农艺性状基因关键功能域氨基酸高密度诱变和定向进化。
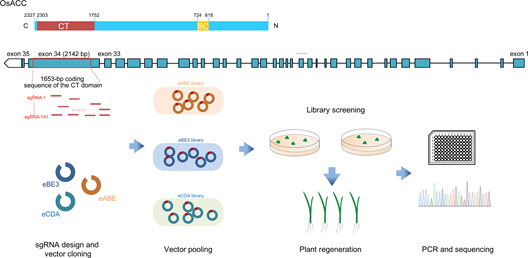
图1, 高密度碱基编辑筛选流程示意图
开发抗除草剂种质材料对规模化作物生产极为重要。ACCase(乙酰辅酶A羧化酶)是芳氧苯氧丙酸(APP)类与环己烯二酮(CHD)类等多种重要商业除草剂的作用位点。许多杂草中, ACCase中羧基转移酶结构域(Carboxyltransferase, CT domain)中某些氨基酸的替换可赋予植物除草剂抗性。为挖掘水稻OsACC抗性位点,创制抗性材料,该研究针对编码OsACC CT domain 的1653bp序列设计了141条sgRNA (NGG PAM),结合该研究组前期创制的两个高效保真CBE工具(eBE3和eCDA,(Qin et al., 2019) )或按照已报道思路优化了前期构建的ABE工具(eABE) (Li et al., 2018; Li et al., 2019),分别构建了三个碱基编辑文库(图1)。理论上,编辑文库的窗口可对CT domain 超1/3的序列实现覆盖(对 eABE, 窗口覆盖611-nt, 含 167 可编辑的 A和 162 T; 对eBE3, 窗口覆盖788-nt, 含165可编辑的C和 192 G; 对eCDA,窗口覆盖393-nt, 含84可编辑的C和92 G)。 在粳稻品种肥粳2020中分别导入三个文库,每文库取5000个转化愈伤在除草剂压力下筛选和分化4~8周后检测所获除草剂抗性事件。 eABE文库共获25个抗性事件,除了一株植物携带I1879V替换外,其他多为前期已报道的C2186R突变(Li et al., 2018); eBE3文库所获三个抗性事件均携带W2125S突变; 而eCDA文库并未获得抗性事件。 进一步结果表明, I1879V突变在水稻正常生命周期中没有体现明显的负面效应, 可能相对较适于进一步的性状改良(图2)。

图2, 不同突变系均表现除草剂抗性
中科院遗传所高彩霞和李家洋老师组近期曾在Nature Biotechnology中报道通过创新构建高效多用途碱基编辑器STEME-1和STEME-NG, 突破PAM限制,实现特定片段饱和的氨基酸突变, 定向进化水稻OsACC基因, 加快了作物性状进化进程(Li et al., 2020)。值得注意的是, 两报道所筛选出的变异并不完全相同。这很大程度上可能由于碱基编辑工具的作用特点、文库的覆盖范围以及筛选方法的不同所造成的。进一步构建更加广适高效的碱基编辑工具, 开发饱和碱基变异筛选方法将极大促进作物重要变异挖掘和性状改良应用。
该工作由安徽省农业科学院和安徽农业大学合作完成, 安徽农业大学农学院吴德祥教授和安徽省农科院水稻所魏鹏程研究员为共同通讯作者。
Reference
Li, C., Zhang, R., Meng, X., Chen, S., Zong, Y., Lu,C., Qiu, J.-L., Chen, Y.-H., Li, J. and Gao, C. (2020) Targeted, randommutagenesis of plant genes with dual cytosine and adenine base editors. Nature Biotechnology.
Li, C., Zong, Y., Wang, Y., Jin, S., Zhang, D., Song,Q., Zhang, R. and Gao, C. (2018) Expanded base editing in rice and wheat usinga Cas9-adenosine deaminase fusion. GenomeBiology 19, 59.
Li, H., Qin, R., Liu, X., Liao, S., Xu, R., Yang, J.and Wei, P. (2019) CRISPR/Cas9-Mediated Adenine Base Editing in Rice Genome. Rice Science 26, 125-128.
Qin, R., Liao, S., Li, J., Li, H., Liu, X., Yang, J.and Wei, P. (2019) Increasing fidelity and efficiency by modifying cytidinebase-editing systems in rice. The CropJournal.
原文链接:
https://onlinelibrary.wiley.com/doi/abs/10.1111/pbi.13348
来源:frontiersin 植物科学最前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247494028&idx=2&sn=79c0d3e434782508a04fb0d7584e4343&chksm=e8bd9b92dfca1284e8554816001960a5bdcaaa32606dc217f805f97dfe8a8e8be1720b1724fb&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

法国法庭禁止出售草甘膦除草剂

各种除草剂的残留毒性有多大?
小麦除草,早上打除草剂好还是下午打除草剂好
不同蔬菜选用不同除草剂

【收藏】除草剂用法最全讲解!
遗传发育所等利用单碱基编辑工具创制抗除草剂小麦

科学家利用单碱基编辑工具创制抗除草剂小麦

推广 | “免费除草一亩地” 傲杀除草剂特别活动
保温栽培蔬菜宜用哪些除草剂?

推广丨工业除草用啥好?“傲杀”帮您解烦恼!