科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-10-22
来源:BioArt
DNA是我们身体里遗传信息的载体。我们每个细胞中的DNA,如果完全伸展开来,是一条长达2米的高分子聚合物。然而,在细胞中,这些DNA需要被储藏在直径只有10微米的细胞核里。我们的细胞通过六百万年的时间进化出了一套精巧的,横跨了六个数量级空间尺度的DNA折叠机制。
染色体拓扑结构域(topologically associated domain,简称TAD)是在亚微米尺度DNA折叠的基本结构单元。TAD中的DNA有更高的概率和区域内而不是区域外的DNA形成物理相互作用。研究显示TAD对基本生物学过程,如细胞分化,转录调控,基因组复制及DNA损伤修复有重要调控作用。
科学界目前尚未对TAD的形成和维持机制达成共识。但是近五到十年来,科学家们提出了一些相关的候选模型。通过染色质免疫共沉淀测序技术,研究发现拓扑结构域的边界富集了特定的分子,因此这些分子被认为与该结构的形成有很大关系。在这些分子中,富集度最高的是转录因子CTCF和蛋白质复合体黏连蛋白复合物 (cohesin)。CTCF有11个锌指结构,能够以非常高的亲和力与DNA结合。Cohesin是一种环状的大分子复合物,它有一个很有趣的特点,就是能够通过消耗ATP在DNA上滑动。根据CTCF和cohesin的这些特点,一些科学家提出了DNA 环外挤 (DNA loop extrusion) 模型用以解释TAD的形成。这种模型预测cohesin在DNA上滑动,在滑动的同时会将穿过其中的DNA外挤形成环状结构,直到遇到稳定结合在DNA上的CTCF之后停止运动。除了CTCF和cohesin,科学家们还发现TAD边界富集有大量常表达基因 (housekeeping gene) 和转运 RNA (tRNA) 基因。因此另一种模型认为活跃的转录和转录相关蛋白,例如RNA聚合酶,与TAD的形成有关。
然而,这些模型,特别是模型涉及到的分子机理,到目前为止还没有得到很好的细胞内实验证据的支持。研究方法的限制是重要原因之一。一方面,我们没有很理想的手段去研究蛋白的特异性作用。传统的控制细胞内蛋白的方法主要是通过mRNA水平的调控,这种方式通常需要较长的时间。由于细胞内的生物化学反应高度关联,且互相影响,不同分子之间形成一个非常密集的调控网络。因此当我们改变这个调控网络的一个节点(某一蛋白)时,这个效果会扩散至整个网络从而影响到其他节点的状态,且等待时间越长,这个效果扩散的范围越大。当我们尝试着去测量下游的结果时,我们实际上测量的并不是单独改变一个节点所带来的效果(原发性效果),而是在这段时间内所有改变节点所带来的效果(我们称其为继发性效果)。因此,由于通过改变mRNA来调节蛋白的水平所需时间较长,我们所测量的结果很多时候是被继发性效果主导的。另一方面,科学家研究TAD的手段大多数还是局限于基因组学的方法,包括染色质免疫共沉淀,以及近年来非常流行的基于染色体构象捕获(chromosome conformation capture,简称3C)的一系列方法。这些方法需要大量的细胞,因此运用这些方法测量出的结果往往是几百万个细胞的平均结果。这种平均结果往往会掩盖描述分子机理的重要物理量,因此理想的方法需要提供单细胞水平的测量能力。
近年来,技术的发展为克服上述技术瓶颈提供了方案。第一项技术是显微镜技术。实际上,相当长时间以来,显微镜学一直是染色质研究的最主要,甚至是唯一手段。显微镜技术的优势之一就是它是单细胞技术,因此自然而然地克服了多细胞平均所带来的问题;显微镜技术的另一大优势,是具备观察活细胞的能力,这是任何的基因组学方法所不具备的。然而,由于光学显微镜技术受限光学衍射极限(约转化成250 纳米的xy分辨率),光学显微技术常年止步于百纳米级别的观察。而TAD的空间尺度恰好就落在这一光学衍射极限附近,因而用传统的显微技术观察起来相当困难。然而超分辨显微镜技术的出现和发展彻底打破了这一窘境(光学超分辨技术获得了2018年的诺贝尔化学奖)。通过巧妙的方法,科学家们开发出了一系列超分辨技术打破了光学衍射极限。这些技术能够获得十纳米级别甚至更高的分辨率。另一方面,近些年飞速发展的基因编辑技术(这项技术的新宠儿,CRISPR/Cas9技术,也刚刚荣获了2020的诺贝尔化学奖)为科学家们提供了一系列强大的工具。通过将基因编辑技术与不同化学生物学方法结合,科学家们现在不但可以特异性的标记内源蛋白,还可以快速精确的控制特定内源蛋白的细胞内浓度。这些新的技术从理论上可以很好的解决之前提到的问题,然而在实验上,特别是在对染色质的研究上,还鲜有应用的例子。
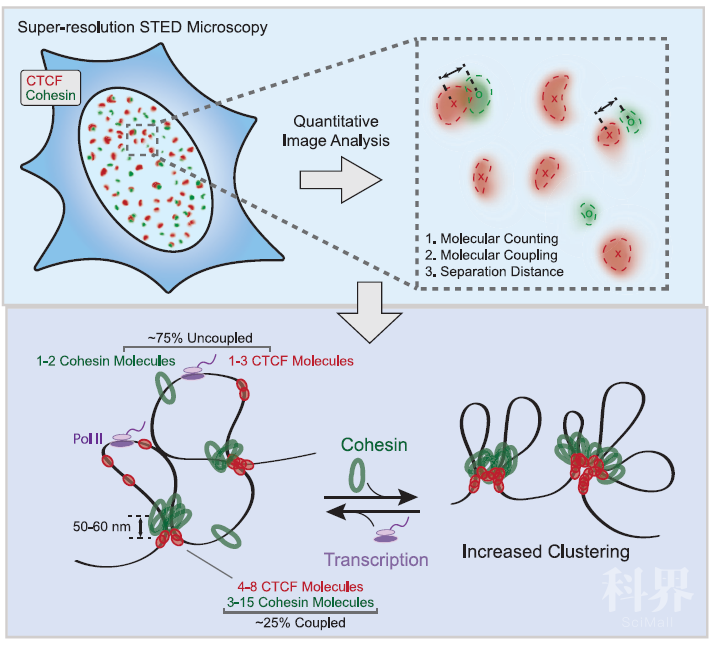
2020年10月21日,斯坦福大学Joanna Wysocka实验室与W. E. Moerner实验室合作(第一作者为古柏博士)在Molecular Cell期刊上发表了题为 Opposing Effects of Cohesin and Transcription on CTCF Organization Revealed by Super-resolution Imaging的文章。在该研究中,作者运用STED(Stimulated Emission Depletion)超分辨光学显微技术以极高的空间(~65 nm)和时间分辨率(1-2 min)定量测量了一系列描述参与TAD形成关键蛋白空间分布的物理量。同时,作者运用CRISPR/Cas9基因编辑技术向内源基因引入了一种叫做degron的多肽。这种多肽能帮助实现该内源基因所编码蛋白的可控的快速降解(1-2 hrs),从而让作者能更精准的了解这些被降解蛋白的特异作用。作者通过结合上述方法,以CTCF蛋白为切入点,在活体单细胞水平,通过分子计数,空间耦合以及空间分布等测量,阐明了该蛋白与cohesin和RNA聚合酶II的空间关系,并且进一步研究了cohesin和基因转录是怎样共同调控CTCF空间分布的。

STED显微技术对于荧光基团的光稳定性有较高的要求。为了能够满足这种要求,作者运用CRISPR/Cas9技术将一种多肽—dL5—插入内源的CTCF分子。通过结合小分子,这种多肽可以发出具有极高光稳定性的荧光。由此,作者运用STED显微技术就可以直接观察内源的CTCF。另外,不同于传统的免疫荧光染色(Immunofluorescence)的方法,由于这种标记内源蛋白的方法可以确保每一个CTCF分子只有一个荧光基团,这一优势能够帮助实现定量的分子计数实验。同时,作者运用了巧妙的生化方法去除细胞中游离CTCF(非DNA结合的)对观察的干扰,因而可以特异性的观察结合在染色质上的CTCF。通过这种方法,作者发现:与平时用传统显微镜观察到的不同,由于空间分辨率的提升,在超分辨显微镜观察下,CTCF呈现出空间分布的异质性,即在染色质上呈现非平滑的空间分布,形成分子团簇。通过分子计数实验,作者发现这些CTCF的分子团簇呈现特定的组成成分。绝大多数的分子团簇含有2-8个CTCF分子。这一测量结果深化了我们对于结合在染色质上的CTCF空间分布的认识。
CTCF与cohesin而非RNA聚合酶II在活细胞中存在空间耦合遵循着类似的思路,作者用两种不同的多肽分别标记内源CTCF,SMC3(cohesin复合物的一个亚基,每个cohesin复合物包含一个SMC3蛋白)和RNA聚合酶II中的任意两种蛋白的组合,并用STED显微技术的不同光谱通道去同时观察两种蛋白在细胞内的空间分布。接着,作者充分利用了显微技术与活细胞兼容的能力,以极高的时间分辨率(最短双通道时间间隔8s)定量测量不同蛋白组合间的空间耦合情况。作者发现,结合在染色质上的CTCF和cohesin在活细胞中存在着显著的空间耦合,而这种空间耦合不存在于CTCF和RNA聚合酶II或者cohesin和RNA聚合酶II之间。这一结果第一次在单个活细胞内验证了CTCF和cohesin存在物理相互作用,同时也为它们功能之间的联系提供了证据。同时,RNA聚合酶II和CTCF以及cohesin的非耦合关系意味着基因转录和DNA环外挤很有可能是两个相对独立的机制。
空间耦合的CTCF和cohesin存在特定的空间排列方式以及组成成分为了能够获得更加定量的测量结果,作者巧妙地借用了应用于另外一类超分辨技术(STORM和PALM)的统计学测量方法,从而以极高的精度(10-15 nm)统计在单个细胞中,耦合的CTCF和cohesin分子所占的比例以及耦合分子间的距离。通过与分子计数实验结合,作者进一步测量了耦合的CTCF和cohesin分子团簇中所含分子的数目。通过以上测量,作者发现单个细胞中大约有25%的CTCF与cohesin空间耦合。同时耦合的CTCF和SMC3分子团簇呈现出特征性的~65 nm的间距,这与cohesin环状结构的直径相似。这一结果首先肯定了与CTCF耦合的cohesin在细胞中以环状结构存在,同时也暗示CTCF和cohesin结合位点不是随机的,这与最近发表的结构学证据高度吻合。另外,通过对比耦合以及非耦合的分子团簇,作者发现与cohesin耦合的CTCF分子团簇通常包含大于四个CTCF分子。反之,与CTCF耦合的cohesin团簇通常包含大于五个cohesin复合物。到目前为止,我们对于DNA环外挤机制的了解绝大多数来源于计算机模拟,然而由于实验数据的匮乏,这些模拟大多依赖假想的物理量。作者通过以上实验测量的物理量可以帮助更好的约束DNA环外挤模型,从而更精准地预测出关于TAD形成的可验证假设。
Cohesin复合物通过其染色质重排作用促进CTCF分子团簇的形成这些CTCF分子团簇形成地机理是什么呢?早期的理论认为,CTCF分子本身有形成寡聚体的倾向,因此这些分子团簇很有可能是CTCF自身寡聚的结果。然而,根据DNA环外挤模型,科学家们提出了另外一个有趣的推测,就是CTCF分子团簇很有可能是cohesin环外挤的一个直接结果。为了直接验证这个推测,作者运用前述的degron系统,通过快速调控染色质结合的cohesin水平,研究cohesin对于CTCF分子团簇的形成的特异性影响。通过快速定量的降解cohesin的染色质卸载蛋白WAPL(即帮助cohesin从染色质上脱离的蛋白),作者可以可逆地增加结合在染色质上的cohesin蛋白量。通过该实验,作者发现在单细胞水平,染色质上结合的cohesin可以特异性地控制CTCF分子团簇的大小,且cohesin越多,CTCF团簇越大。而且CTCF团簇的增大伴随着CTCF与cohesin空间耦合的增加。同时,cohesin的这种调控机制并不是通过简单地控制CTCF在染色质上的结合总量而实现的。通过以上结果,作者推断cohesin通过其环外挤活性,调控CTCF分子团簇的形成。结合之前的关于耦合CTCF和cohesin团簇地定量分析,这一结果不但说明了CTCF分子团簇的形成特异性地依赖于cohesin,而且为DNA环外挤模型提供了有力的细胞内实验证据。
Cohesin和基因转录相互拮抗调控CTCF分子团簇的形成
根据WAPL降解实验的结果,作者推断如果降解cohesin复合物本身,CTCF分子团簇应该会缩小。然而,令作者吃惊的是,当作者用degron系统降解SMC3时,CTCF分子团簇的大小并没有改变。这说明细胞内很可能还存在其他的机理去平衡cohesin对于CTCF分子团簇的调控,从而能够在正常情况下,维持CTCF分子团簇在一个较低的水平。由于基因转录和TAD之间的紧密联系,作者猜测这种平衡机制很有可能是基因转录。为了验证这个假设,作者利用小分子去特异性的抑制基因转录,发现CTCF分子团簇在基因转录被抑制时增大,而如果同时降解cohesin复合物,CTCF分子团簇的改变会被逆转。这一结果不但从另一个侧面证明了cohesin能够调控CTCF分子团簇的形成,而且证明基因转录的确作为一种平衡机制与cohesin相互拮抗调控CTCF分子团簇在细胞内的水平。
总结起来,这项研究定量的描述了参与TAD形成的重要蛋白,包括CTCF,cohesin和RNA聚合酶II在染色质上的空间分布和分子成分。通过这些测量,该研究不但定量研究了CTCF分子团簇这一重要现象,同时揭示了cohesin和基因转录对CTCF分子团簇的共同调控。研究发现的CTCF分子团簇的调控机制不但首次为DNA环外挤提供了细胞内证据,而且还从新的角度为基因转录和DNA环外挤机制对TAD形成的共同调控提供了可能的分子机理。最后,这项研究中所使用的实验方法具有很强的普适性,因此这套方法不但可以用于量化研究其他染色质相关分子间相互作用,还可以轻易的被运用到细胞内其它系统的研究中。
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652504516&idx=7&sn=cb3dd5b4148cfe5289eea13c5245116d&chksm=84e19270b3961b66a5b22c1bf232e1df8b1d5b0c6cd39efe2b3189ea86f3624bc60a1f5d39e0&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

Cell : 破解异染色质细胞核周定位之谜
论文导读——染色质重排、染色体破碎、线粒体DNA变化等
Cell丨抑制性染色质如何调控基因的转录?
非对称聚合物材料可制造灵活多变的软机器人
合成生物学: 一个用来控制转基因生物的内置毁灭开关
X染色质
Cell :朊蛋白介导激活型染色质的遗传

自然中不存在的蛋白质也能人造了!撼动材料医药界

DNA机器人一小步 癌症治疗一大步

夫妻相真的存在,研究发现夫妻DNA相似度高过陌生人