科技工作者之家
科界APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-03-13
内容来源:中国科学院
DNA 6mA(N6-甲基腺苷)作为DNA的第二种修饰形式,是哺乳动物基因组表观遗传调控的重要组成。基因组6mA的水平在生物体内具有调节组织发育、性别比例、基因表达、X染色体失活等多种作用,阐明其调控机制是解码这一新型修饰碱基生物学功能的关键。2016年,耶鲁大学Andrew Xiao首次报道ALKBH1在真核生物中具有DNA 6mA去甲基化酶活性,而后一系列的研究表明人源ALKBH1的异常表达导致了多种发育缺陷和癌症的发生。然而,ALKBH1的生化机理和分子机制并不清楚。
3月10日,Cell Research 正式发表了中国科学院生物物理研究所许文青/梁栋材课题组与中国农业大学陈忠周课题组合作的题为Structural basis of nucleic acid recognition and 6mA demethylation by human ALKBH1 的研究论文,在ALKBH1的核酸底物识别及6mA去甲基化机制研究方面取得进展。
该工作首次报道了人源ALKBH1 free状态以及ALKBH1-Mn2+-α-KG复合物的高分辨率晶体三维结构。ALKBH1区别于AlkB家族其它蛋白,具有多个新的结构特征。其独有的Flip0区域,位于活性中心的口袋附近,对于ALKBH1底物识别和发挥6mA去甲基化酶活性中起着至关重要的作用。远离活性中心且带正电荷的Flip1,在面向活性中心的一侧有大量的碱性氨基酸残基,形成一个较大的正电荷区域,这些碱性残基的突变使ALKBH1丧失了与DNA的结合能力。此外,ALKBH1结合α-KG后活性中心附近的Tyr184和Glu236向活性中心移动并通过氢键与Arg344形成稳定的三角形结构,保证了ALKBH1 执行6mA去甲基化酶的活性。此外,在ALKBH1结构中Flip1、Flip2含有很长一段Loop,其构象变化与位于Flip2上的Tyr177、Trp179一起参与了DNA 6mA的识别和结合。在这个工作中,研究人员还发展了一种新颖、稳定且高通量的6mA去甲基化的体外酶促方法。通过这个方法对不同底物进行初筛,结合三维结构分析和质谱测定结果表明,ALKBH1的优先底物是凸起状或气泡状的非配对dsDNA,Bulge或Bubble;而不是ssDNA或dsDNA。这种对局部不配对特征的核酸修饰碱基去甲基化在生理学上有重要意义。这项研究工作为ALKBH1的底物选择机制、催化反应机理和生理功能的探究提供了结构基础,并为相关疾病的药物设计提供了重要依据。
生物物理所许文青/梁栋材课题组副研究员闫小雪和中国农业大学陈忠周为论文的共同通讯作者,博士研究生田利飞和助研刘晏平为论文的共同第一作者,博士陈联琦、唐群和孙伟,以及中国农大博士吴薇参与了该项工作的完成。上海同步辐射光源(BL19U1、BL17U1)和生物物理所科学研究平台为该研究提供了重要的技术支持。这项研究得到中科院战略性先导科技专项、生物大分子国家重点实验室和国家自然科学基金的资助。
论文链接
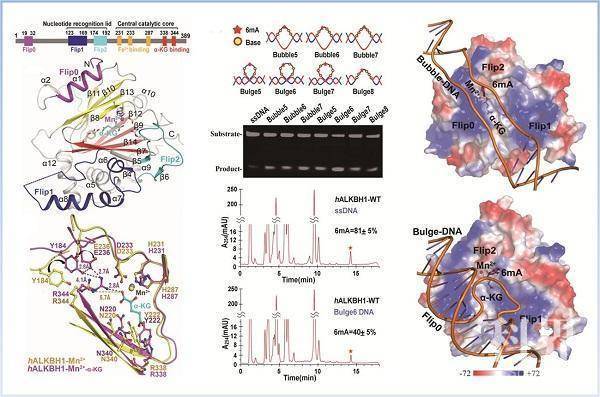
ALKBH1识别与催化Bubble/Bulge DNA 6mA去甲基化的结构基础
来源:中国科学院
原文链接:http://www.cas.cn/syky/202003/t20200312_4737541.shtml
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
TtCas1-DNA复合物的结构生物学研究

中国学者首次揭示ALKBH1作为未配对DNA的N6-mA去甲基化酶

Nature :DNA结构的发现:开创生物学研究新纪元
研究发现DNA甲基化修饰精准调控植物生物钟周期

中国科学院水生生物研究所关于广布性冷水鱼类温度适应机制研究取得新进展

【海洋科普】基瓦多毛怪

人工细胞与生物细胞首次实现成功融合与协作

人体细胞内存在“铁路”系统,能自动优化结构
研究人员在大麻素受体的结构生物学研究中获重要成果

中国海洋湖沼学会理事张奇研究员主持的《鄱阳湖水文情势变化研究》出版发行