科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-08-25
来源:BioArt
染色质构象捕获技术(3C和Hi-C)及其衍生技术(DNase Hi-C和Micro-C )发现并解析出染色体在不同层级上、不同分辨率下的折叠形式。现有的染色质构象捕获技术依靠甲醛交联蛋白与DNA,通过限制性内切酶或者DNase, Mnase对DNA进行酶切,再将空间上邻近的DNA重新连接。将重连后的DNA文库通过高通量测序,分析比对其来源并绘制染色质互作图谱。通过这种互作图人们发现多种染色体折叠形成的结构,其中包括染色质环【1】、染色质结构性条纹(stripe)、染色质结构域、拓扑结构域【2】以及活跃与失活的区室【3】。
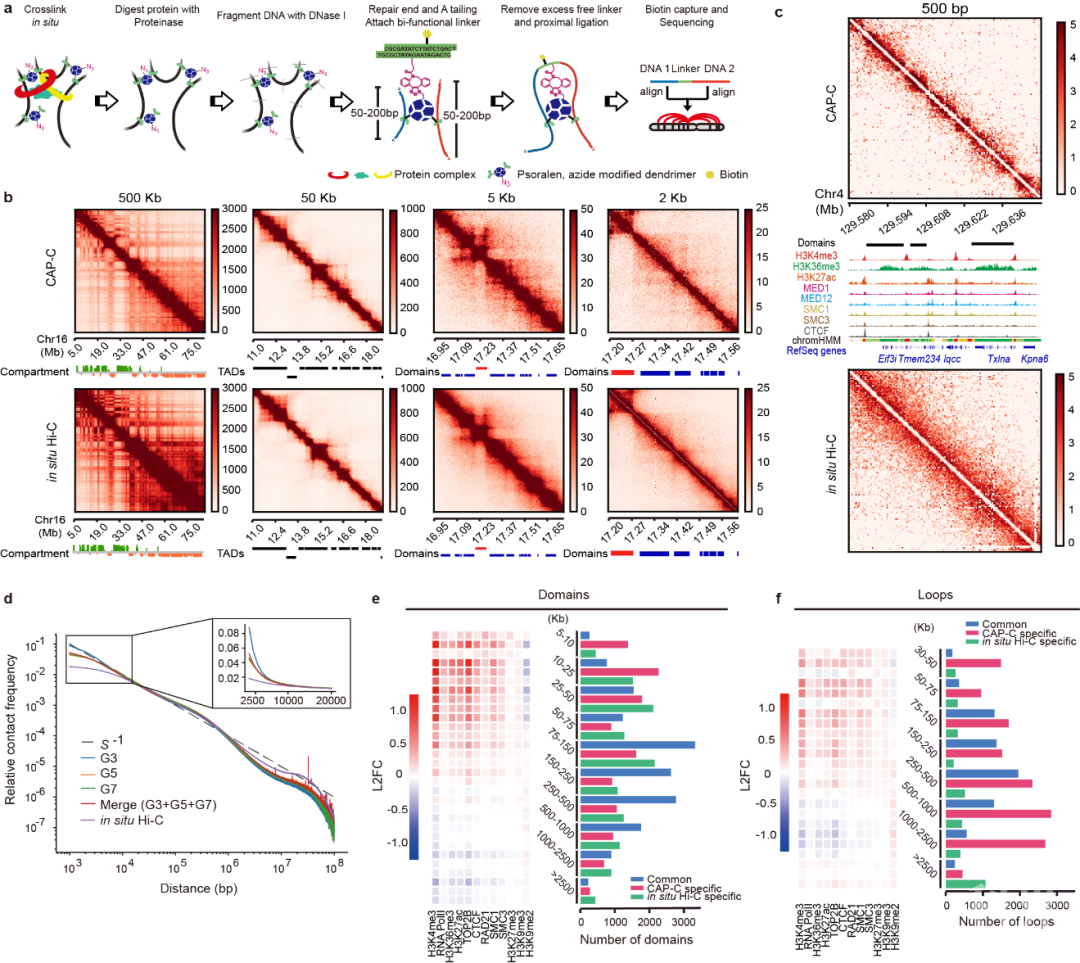 图1. CAP-C实验设计。CAP-C能有效提高染色质互作图上的“信噪比”,帮助发现更多染色质精细结构。
图1. CAP-C实验设计。CAP-C能有效提高染色质互作图上的“信噪比”,帮助发现更多染色质精细结构。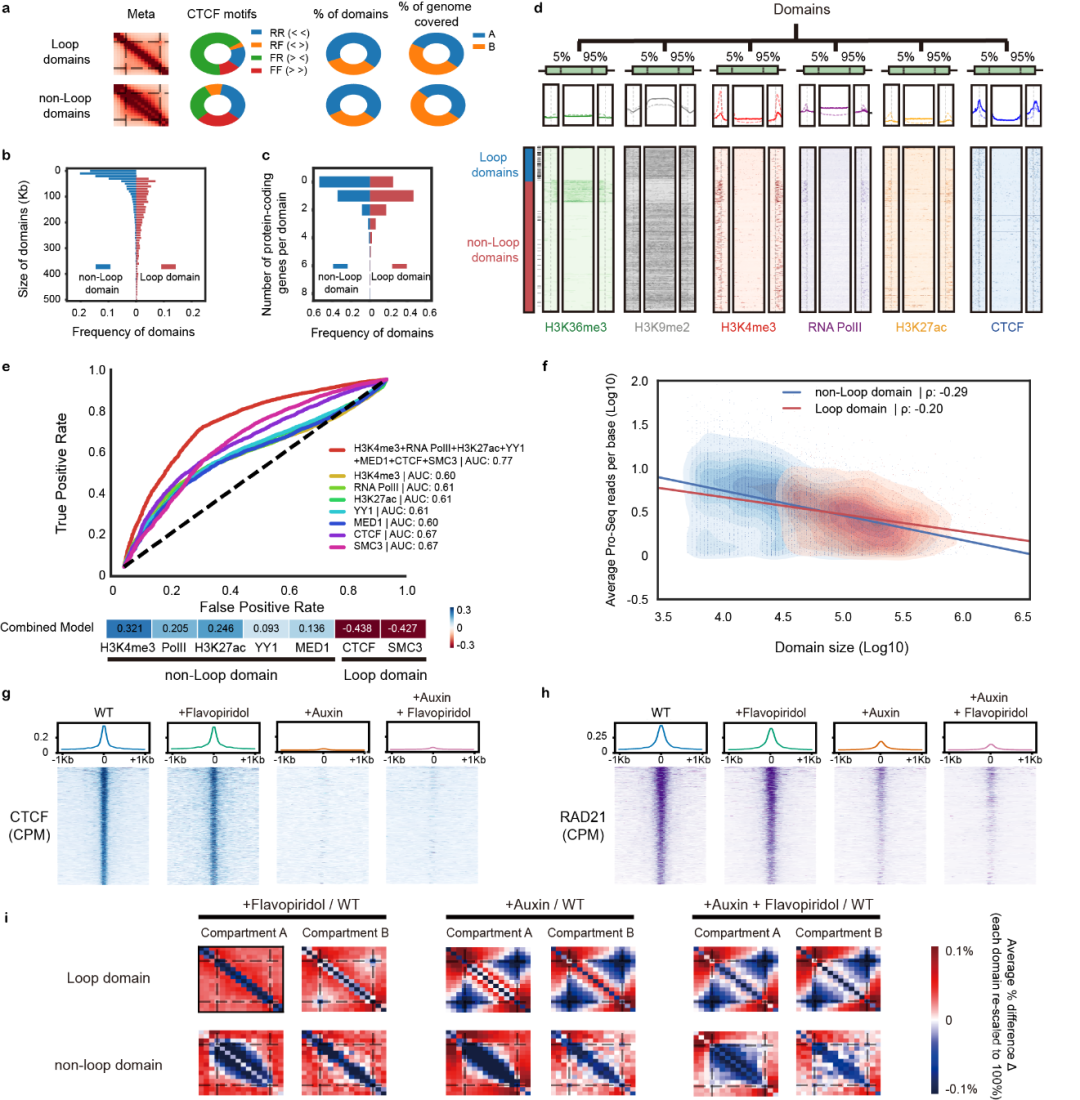 图2. 环状结构域和非环状结构域的对比。
图2. 环状结构域和非环状结构域的对比。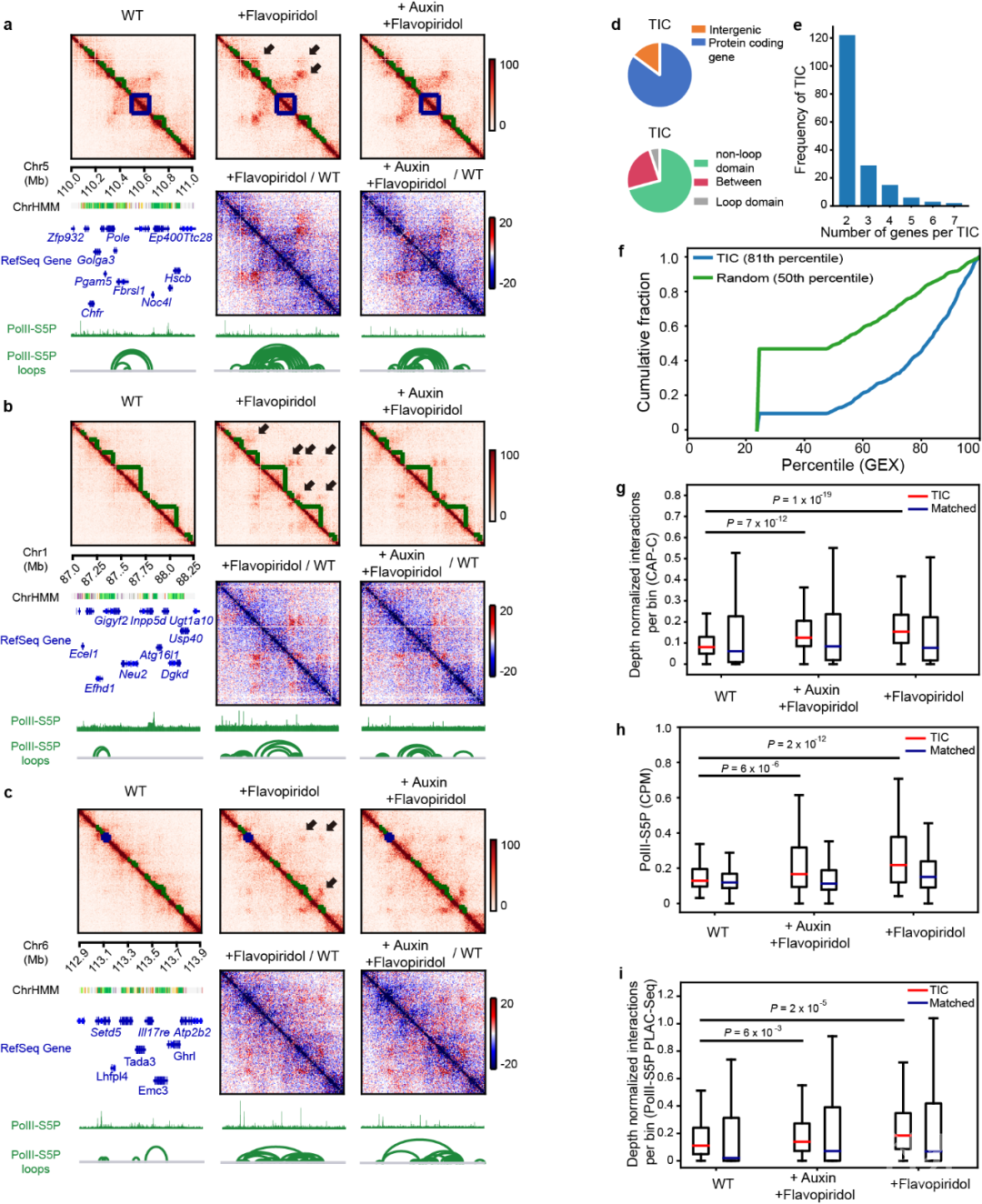 图3. 转录起始簇
图3. 转录起始簇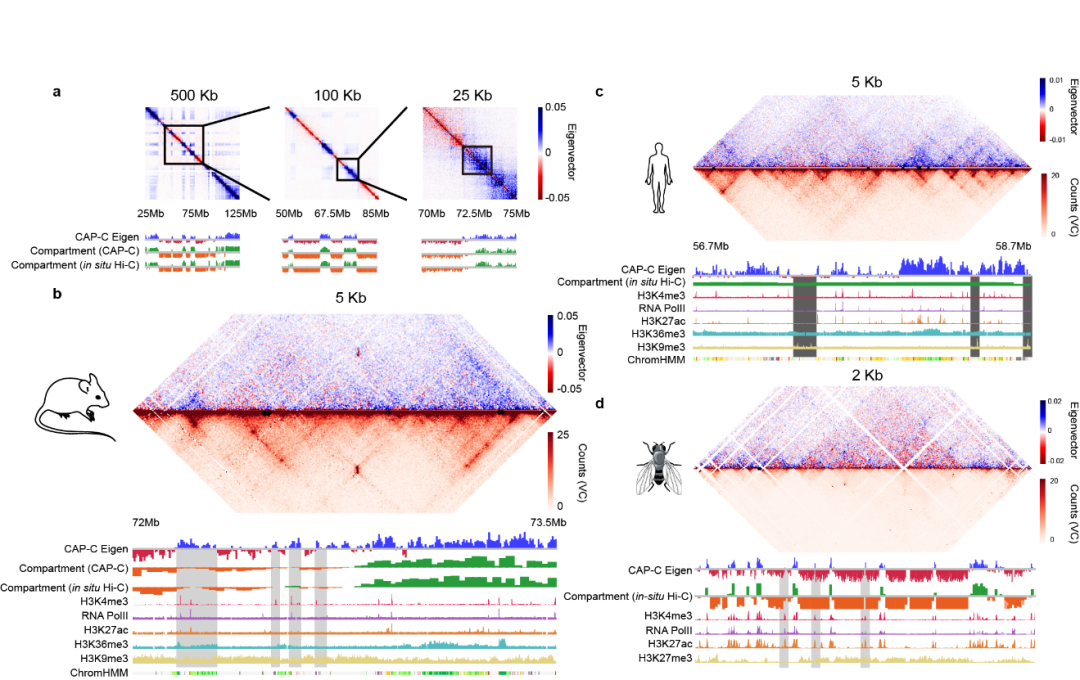 图4. 精细的区室结构存在于各类物种中。图中,染色质被小树枝状高分子(G3)更多的捕获的区域用红色标注,染色质被大树枝状高分子(G5或G7)更多的捕获的区域用蓝色标注。
图4. 精细的区室结构存在于各类物种中。图中,染色质被小树枝状高分子(G3)更多的捕获的区域用红色标注,染色质被大树枝状高分子(G5或G7)更多的捕获的区域用蓝色标注。来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652496965&idx=1&sn=39a4359abece8c7214b3f1a15597bbd7&chksm=84e277f1b395fee725ac2c6826ebeef634290cc9dc71fddba13f89e6669970948e5a1649c186#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

SWI/SNF染色质重塑因子突变与癌症
论文导读——染色质重排、染色体破碎、线粒体DNA变化等

朱健康团队综述 | 化繁为简,植物多基因编辑体系的优化

Nature | 异染色质空间隔离调控新思路

北京基因组所开发出在单细胞中识别染色质类染色质拓扑相关结构域结构的算法

苹果着色谁说了算?科学家有新发现

全面分析拟南芥、玉米和高粱核心启动子,助力设计新启动子和作物改良
植物染色质三维构象的深度解读
Cell :朊蛋白介导激活型染色质的遗传
X染色质