科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-07-16
来源:BioArt
RNA 不仅起到传递遗传信息、翻译蛋白的作用,在细胞内还发挥非常广泛和关键的调节功能, 如小RNA和非编码RNA等【1】。而RNA功能主要是由其核苷酸序列决定的,特别是5'和3'末端序列对其功能至关重要【2】。在细菌体内,RNA 的转录、翻译和降解常常同时进行,每个RNA分子的5'和3'末端动态变化是体内一系列转录调节的产物【3】。因此,测定所有RNA分子的全长序列可以更好地了解细胞功能、基因调节网络,特别是同RNA 有关机制的相互作用。
新一代高通量RNA测序是用于分析转录组学革命性的工具【4】。基于Illumina 平台的高通量测序可以轻易地得到大量的测序短序列,但通常需要将RNA进行打断,从而失去了RNA的完整序列信息;而基于PacBio平台的长序列测序可以得到RNA完整序列信息,但目前仍不能满足转录组研究的准确性、高深度和高可靠性的要求。因此一种能够结合两者优势的方法必将深受欢迎。
2019年7月17日,洛克菲勒大学刘诗欣实验室(第一作者为鞠湘武博士)在Nature Microbiology发表文章Full-length RNA profiling reveals pervasive bidirectional transcription terminators in bacteria,报道了他们研究的一种新的RNA测序方法,5'和3'末端连同测序(simultaneous 5' and 3' end sequencing, SEnd-seq)。

这种方法通过将RNA末端标记后,逆转录成单链cDNA,然后将cDNA进行环化,使得cDNA的两个末端连接在一起。随后将环化的cDNA 打断后,将同时包含RNA 5’端和3’端的片段进行富集并制备成测序文库,在Illumina 平台如Next-seq 500进行测序。通过相应的生物信息学分析可以精确地还原RNA的相应末端信息。由于在细菌中内含子并不常见,因些可以根据两处末端信息推断出每个RNA分子的完整序列信息。
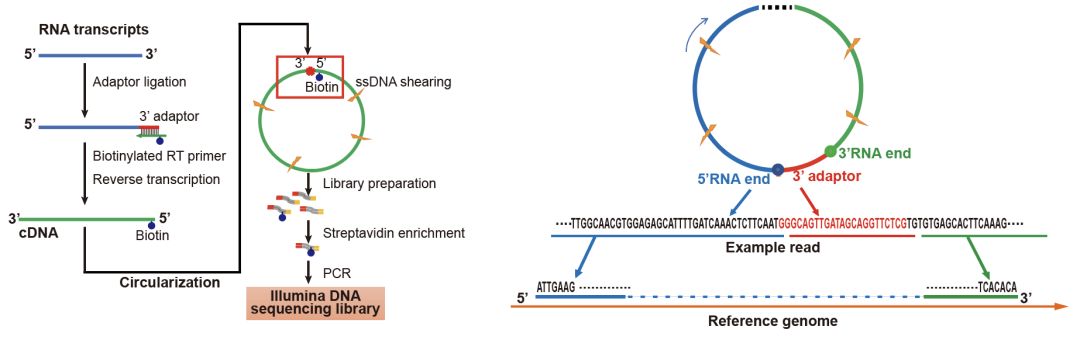
图1:SEnd-seq RNA测序流程
该论文使用了一种高通读性的II型内含子成熟酶,来尽可能地将长度各异、包含各种复杂二级结构的全长RNA完整地逆转录成cDNA;并通过优化反应条件,使得TS2126连接酶能高效地将不同长度的cDNA环化。文章在随后一系列的质量控制分析中,显示SEnd-seq并没有引起明显的长度或者末端序列偏差,也几乎没有检测到不同cDNA的线性连接,而且得到基因表达水平同普通RNA测序结果高度相关。
因此通过SEnd-seq RNA测序方法,该文作者绘制出精确到单个核甘酸的高分辨率大肠杆菌转录组图谱。同时结合相应方法,SEnd-seq可以在大肠杆菌中检测出比以前更多的转录起始位点 (TSS) 和转录转录终止位点(TTS),并成功注释大部分转录终止位点类型如固有终止子(Intrinsic terminator)和Rho蛋白依赖的终止子(Rho-dependent terminator)。而且SEnd-seq方法可以非常直观地显示转录组操纵子结构,反义RNA分布等多种信息。
除此之外,他们还发现很多两个转录会聚方向的RNA转录物经常会出现3’末端重叠,从而形成双向转录终止子(Bidirectional terminator)。而这两个转录本可能是由一对转录相反方向的基因或基因和反义编码RNA之间组成。但大部分双向转录终止子并不是两个转录终止子的简单重叠,提示其它的机制可能在发挥作用。通过体外和体内的实验进一步证明,两个会聚行进方向的RNA 聚合酶在双向转录终止子附近的碰撞会引起很高的转录终止效率,比仅仅单一方向的转录终止效率要高得多。这一发现进一步将基因转录排列方向同转录功能及产物联系起来。
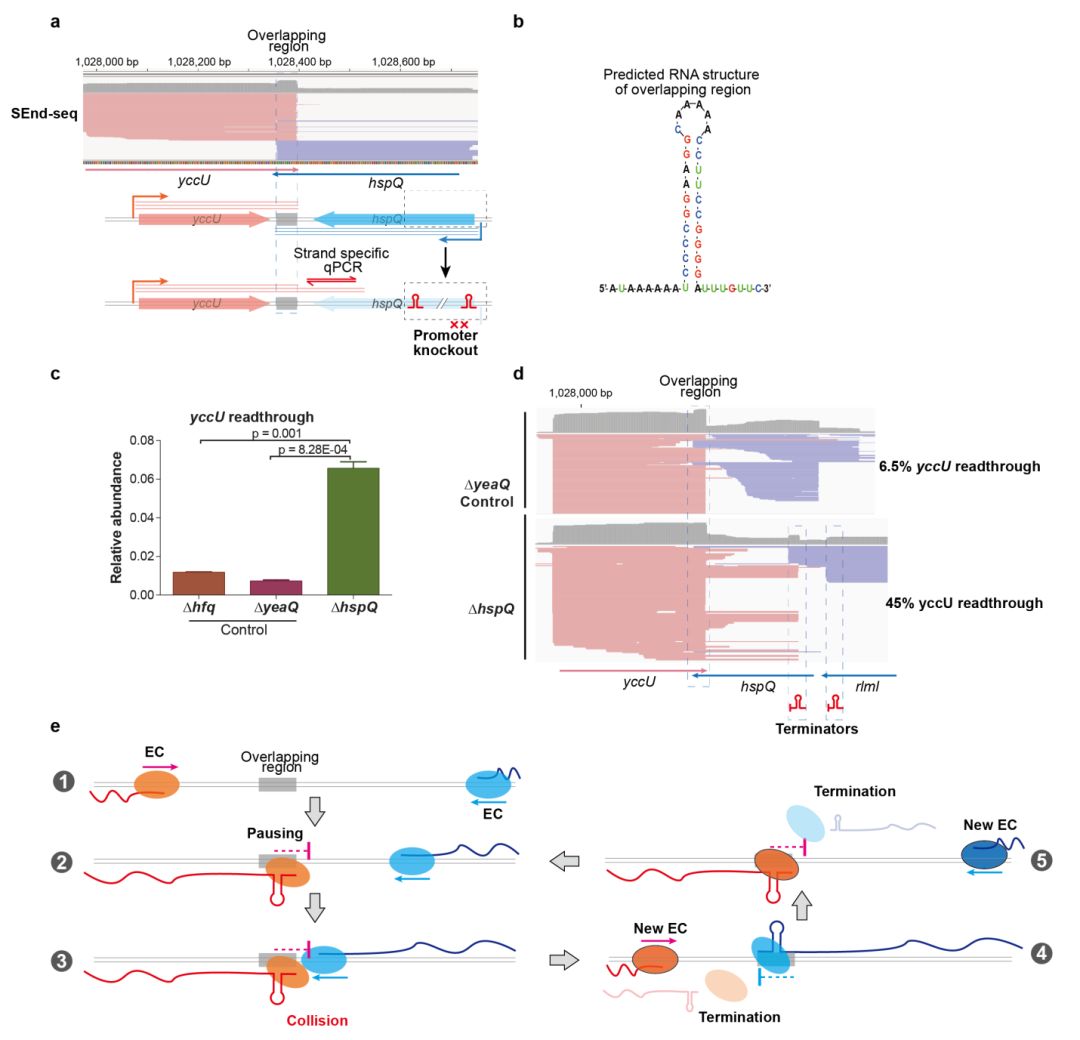
图2:会聚行进方向的RNA 聚合酶碰撞引起转录终止示意图
以上结果表明,SEnd-seq是一种功能强大的测序方法。而且一对测序序列可以对应一条RNA分子,因此很容易得到很深的测序深度并节省测序费用。综上, SEnd-seq完全可以进一步被广泛地应用于病原细菌转录组和亚转录组等方向的研究。
原文链接:
https://doi.org/10.1038/s41564-019-0500-z
制版人:珂
参考文献
1. Morris, K.V. & Mattick, J.S. The rise of regulatory RNA. Nat Rev Genet 15, 423-37 (2014).
2. Davuluri, R.V., Suzuki, Y., Sugano, S., Plass, C. & Huang, T.H. The functional consequences of alternative promoter use in mammalian genomes. Trends Genet 24, 167-77 (2008).
3. Hor, J., Gorski, S.A. & Vogel, J. Bacterial RNA Biology on a Genome Scale. Mol Cell 70, 785-799 (2018).
4. Wang, Z., Gerstein, M. & Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet 10, 57-63 (2009).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652472589&idx=4&sn=c1603ec9ee71d932f4265015672279ad&chksm=84e216b9b3959faf8e0c010b34b9b0b443521205ac9ca3b543531fa7c530a3c56998eaab46bc&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
MPB:沈阳生态所李琪组-土壤线虫群落DNA提取、扩增及高通量测序

中华预防医学会关于对《基于高通量测序的病原体筛查通用准则》团体标准征求意见稿公开征求意见的通知

Nat Protocols | 陈亮/付向东合作组报道R-loop的新测序技术

在一天内完成样本制备到结果报告全流程的高通量基因测序平台
浙大刘建钊、北大贾桂芳团队分别开发新的m6A高通量测序方法

Cell:高质量&高通量!马赛博士等开发单细胞测序新技术测定染色质潜力
JIPB:赖锦盛课题组建立一种适用于高通量转录组和基因型分析的测序文库构建方法

程溪柳/刘君/库丽霞合作开发TEAseq技术,用于高通量测序鉴定玉米转座子插入突变体库
肿瘤基因检测小知识 | 液态活检之循环肿瘤DNA

学术发布 | 促进宏基因组高通量测序技术在感染性疾病病原检测中的规范应用